Delavnica: MolStar#
Namen#
Namen delavnice je seznaniti se s sodobnim in enim najpogosteje uporabljanim orodjem za vizualizacijo bioloških makromolekul v brskalniku oz. na spletu – programom MolStar, ki ga za vizualizacijo struktur uporabljata med drugim tudi zbirke RCSB PDB, PDBe in AlphaFold Protein Structure Database.
Potrebščine#
Za izvedbo potrebujete:
spletni brskalnik (najbolje na namiznem ali prenosnem računalniku, pogojno na tablici, na slednji je sicer malce težje manipulirati z modeli struktur; mobilni telefon ni primeren),
povezavo v splet.
Miška
Priporočam uporabo miške, ki omogoča levi in desni klik ter kolešček (sredinski klik), saj to bistveno olajša manipulacijo struktur v 3D.
Shema delavnice#
Na delavnici bodo predstavljeni spodaj navedeni vidiki uporabe orodja MolStar. Delavnico ste uspešno opravili če lahko za vsako od navedenih točk rečete “Vem, za kaj se gre in znam uporabiti!”.
nalaganje datotek iz zbirke PDB – na primeru človeškega citokroma P450 (PDB ID 1TQN)
podrobnejši pogled na določen del strukture (focus)
preko klikanja po zaporedju
spreminjanje prikaza aminokislinskega zaporedja, ligandov, vode, …
preko klikanja na strukturo
ponastavitev pogleda
tip prikaza strukture (Model, Assembly, Symmetry Mates) – to se pojavlja le pri nekaterih strukturah (odvisno od metode določitve)
spreminjanje prikaza osnovnih komponent
hitri stili (Quick Styles)
predpripravljeni načini prikaza (Preset)
vklop/izklop prikaza posameznih komponent
spreminjanje načina prikaza komponent (različni tipi prikaza, barvanje)
prikaz oznak ak ostankov
izbiranje posameznih ak ostankov oz. delov strukture
spreminjanje načina prikaza izbranih delov modela
ustvarjanje novih komponent ter dodajanje načinov njihovega prikaza
prikaz nekovalentnih interakcij
meritve razdalj
superpozicija (superpozicija z govejim citokromom P540 (PDB ID 3MZS))
nastavitve vizualizacije
barva ozadja
prikaz režnja (clipping)
pogled iz perspektive
shranjevanje slik in filmov
slike
filmi oz. animacije
shranjevanje in ponovno nalaganje sej
primerjava med samostojnim MolStar (https://molstar.org/viewer/) ter MolStar, integriranim v RCSB PDB
nalaganje lokalnih datotek in shranjevanje sej (omejeno v MolStar @ RCSB PDB)
hierarhičen prikaz odprtih struktur (samo v samostojnem MolStar)
(manjkajoča “leva” kontrolna plošča v MolStar @ RCSB PDB)
3D kartiranje pripisov na strukturo (samo v MolStar @ RCSB PDB)
Poizkusite sami#
Učite se iz primerov
Vse interaktivne vizualizacije molekul, ki so pripravljene v tej spletni knjigi in jih uporabljamo med predavanji, so prav tako ustvarjene v programu MolStar. Privzeto so pri teh prikazih kontrolne plošče skrite, lahko pa si jih prikažete s klikom na simbol za orodni ključ () in analizirate seje v smislu komponent in načina njihovega prikaza.
Dodatni primeri#
Spodaj je prikazanih nekaj primerov, ki jih lahko poizkusite poustvariti – vsak je opisan, dodana je tudi slika, kako bi naj vaš prikaz na koncu izgledal, vsak primer pa ima dodano tudi povezavo do seje, uporabljene za ustvarjanje te slike.
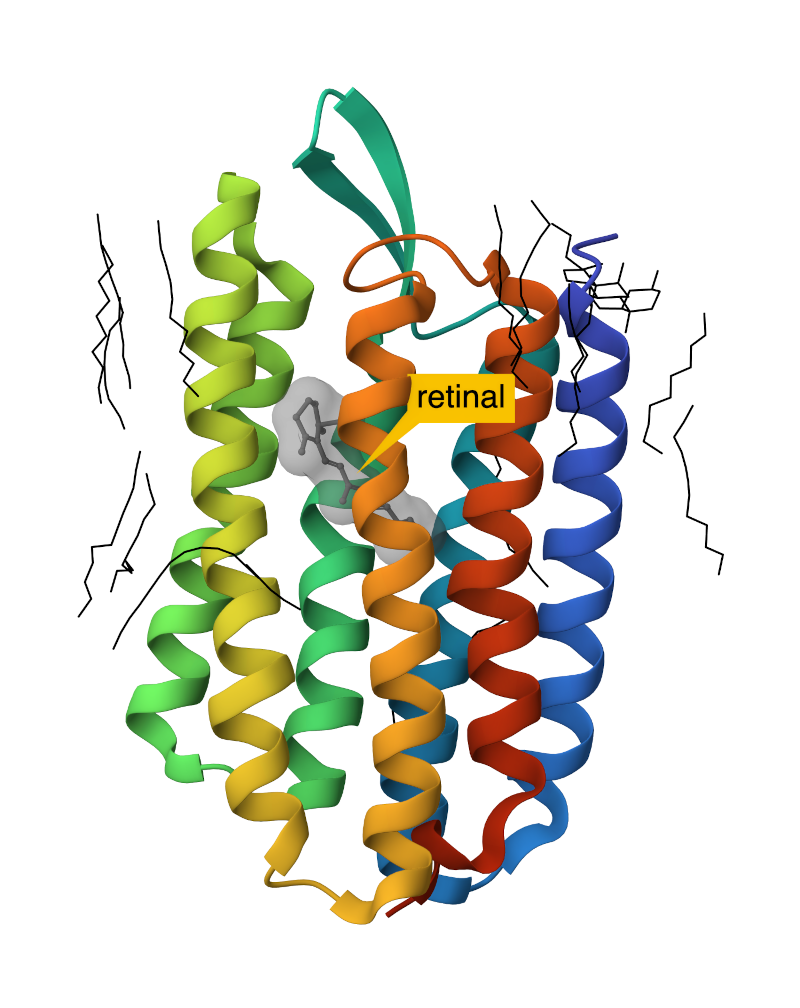
Model strukture mutiranega bakteriorodopsina iz Halobacterium salinarum (PDB ID 3HAP):
protein kot mavrično obarvan trak (N-konec moder, C-konec rdeč),
retinal na način kroglice in paličice, ogljikovi atomi naj bodo sive barve, dodana pa naj bo prosojna (50 % prosojnost) sivo obarvana molekulska površina,
retinal naj bo posebej označen kot ‘retinal’ z rumenim ozadjem, oznaka pa naj bo malce oddaljena od liganda in do njega povezana s puščico,
molekule detergenta in druge molekule, ki so sestavljale bicele, kot črno obarvane odebeljene črte,
voda ni prikazana.
Rešitev
Na voljo je seja, uporabljena za pripravo slike, prikazane zgoraj.
Namig: za povezovanje oznake z nekim delom modela morate vključiti Tether pod naprednimi nastavitvami oznake (Advanced Options).
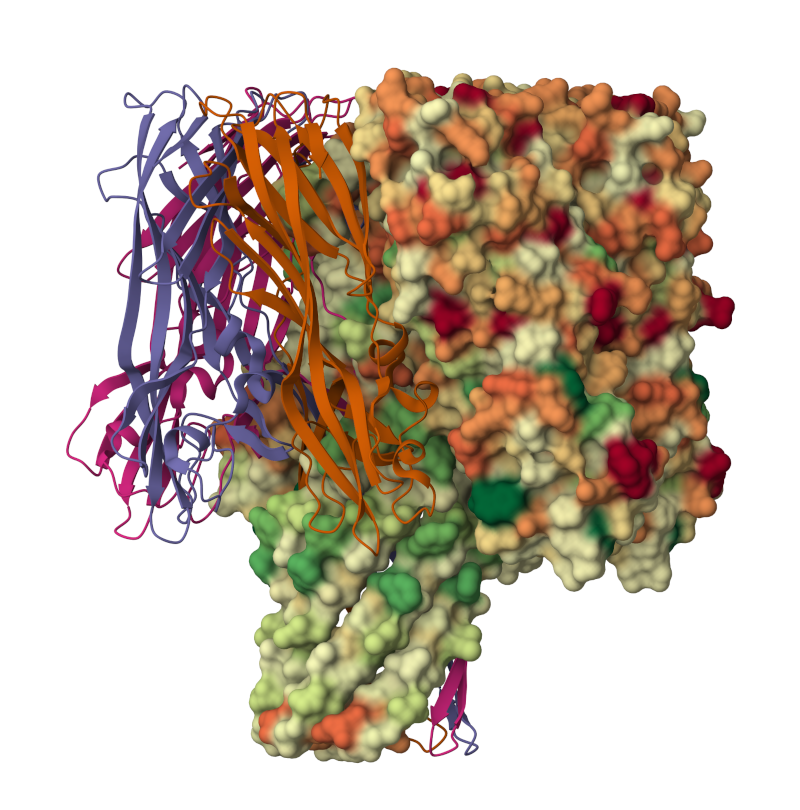
Model strukture mutiranega \(\alpha\)-hemolozina iz Staphylococcus aureus (PDB ID 3M2L):
3 podenote prikazane kot trak različne barve,
4 podenote prikazane kot molekulska površina, obarvana glede na hidrofobnost,
voda ni prikazana
Rešitev
Na voljo je seja, uporabljena za pripravo slike, prikazane zgoraj.
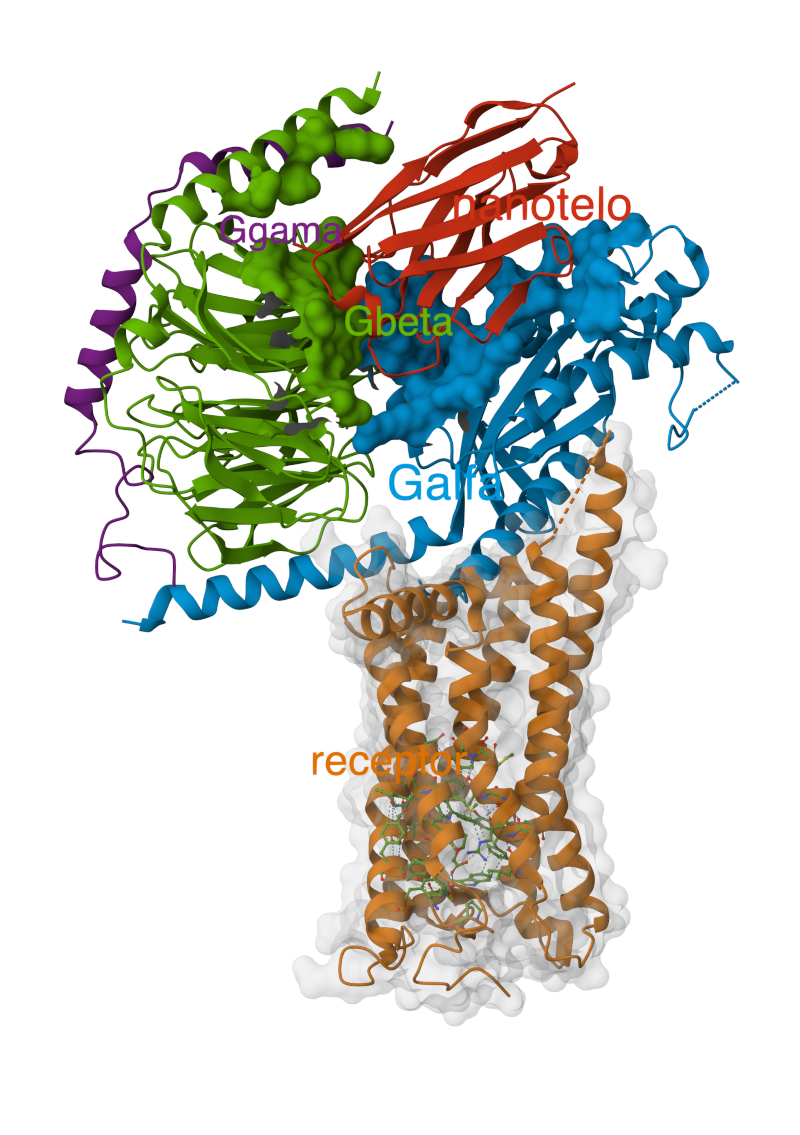
Model strukture kompleksa med transmembranskim receptorjem, v katerega je vezan ligand MRE (aktivna oblika spojine selexipag), heterotrimernim kompleksom G proteinov mutiranega alfa-hemolozina iz Homo sapiens ter nanotelesom (PDB ID 8X79):
nanotelo prikazano kot trak rdeče barve,
Gs\(\alpha\) kot trak modre barve,
G\(\beta\) kot trak zelene barve,
G\(\gamma\) kot trak temno vijolične barve,
aminokislinski ostanki G proteinov, ki so od nanotelesa oddaljeni največ 5 Å, kot površina (ampak zgolj kot del zunanje površine proteina!),
receptor kot trak oranžne barve, dodana naj bo prosojna molekulska površina sive barve (75 % prosojnost),
ligand MRE kot kroglice in paličice,
prikaz vodikovih atomov naj bo izključen,
aminokislinski ostanki receptorja, ki so v stiku z ligandom MRE, kot kroglice in paličice, vključen pa naj bo tudi prikaz vseh nekovalentnih interakcij med temi aminokislinskimi ostanki in ligandom,
vse proteinske verige naj bodo označene (receptor, Galfa, …), barve oznak naj bodo enake kot so barve verig, obroba oznak naj bo skrita.
Rešitev
Na voljo je seja, uporabljena za pripravo slike, prikazane zgoraj.
Namigi:
Da dobite pravilen prikaz interakcijske površine med nanotelesom in ostalimi polipeptidnimi verigami morate pri naprednih nastavitvah (Advanced Options) dotične molekulske površine vključiti Ignore Parent.
Poigrati se morate z oznakami in jim nastaviti mesto pritrditve (Attachment) ter odmik (Offset), da bodo bolje vidne.
Nekatere oznake bodo med vrtenjem izginjale, to “popravite” tako, da nastavite Clipping na 0.