Kratka navodila
Pri delu bomo uporabljali program Moorhen, ki predstavlja spletno različico programa COOT (Crystallographic Object-Oriented Toolkit), danes najpogosteje uporabljanega programa za gradnjo, piljenje in validacijo modelov struktur makromolekul [Emsley et al., 2010]. Isti program se lahko uporablja tako za gradnjo modelov struktur na osnovi podatkov, pridobljenih z difrakcijo, kot tudi s krio-elektronsko mikroskopijo.
Začetni podatki
Za demonstracijo gradnje in piljenja modela bomo uporabili skoraj popolnoma zgrajen in izpiljen model strukuture človeškega estrogenskemu receptorju podobnega receptorja gama (ESRRG, UniProt P62508).
Aminokislinsko zaporedje kristaliziranega konstrukta je podano spodaj:
LGSPEFLNPQLVQPAKKPYNKIVSHLLVAEPEKIYAMPDPTVPDSDIKAL
TTLCDLADRELVVIIGWAKHIPGFSTLSLADQMSLLQSAWMEILILGVVY
RSLSFEDELVYADDYIMDEDQSKLAGLLDLNNAILQLVKKYKSMKLEKEE
FVTLKAIALANSDSMHIEDVEAVQKLQDVLHEALQDYEAGQHMEDPRRAG
KMLMTLPLLRQTSTKAVQHFYNIKLEGKVPMHKLFLEMLEAKVC
N-končni del zaporedja LGSPEF in C-končni ostanek C niso del zaporedja v UniProt, tako da le regija L220–V458 ustreza zaporedju pod zgoraj omenjenim UniProt zapisom.
Drugi relevantni podatki:
kristalizacijski pufer: 0,05 M HEPES, 0,75 M natrijev citrat, 5 % glicerol, pH 7,5
kokristalizacija z bisfenolom A
ločljivost: 1,6 Å
prostorska skupina: P 41 21 2 (tetragonalna), 8 asimetričnih enot na osnovno celico
Povezavi za prenos datotek, potrebnih za delo:
Gradnja in piljenje modela
Nalaganje podatkov
Običite stran moorhen.org. Program je še v razvoju, zato se lahko delovanje v nekem časovnem obdobju spremeni.
Naložite podatke:
Povežite model strukture s strukturnimi faktorji in s tem omogočite osveževanje karte elektronske gostote v realnem času ter izračun faktorja R: Menu > File > Connect mol. and map for updating… > (preverite nastavitve) > OK
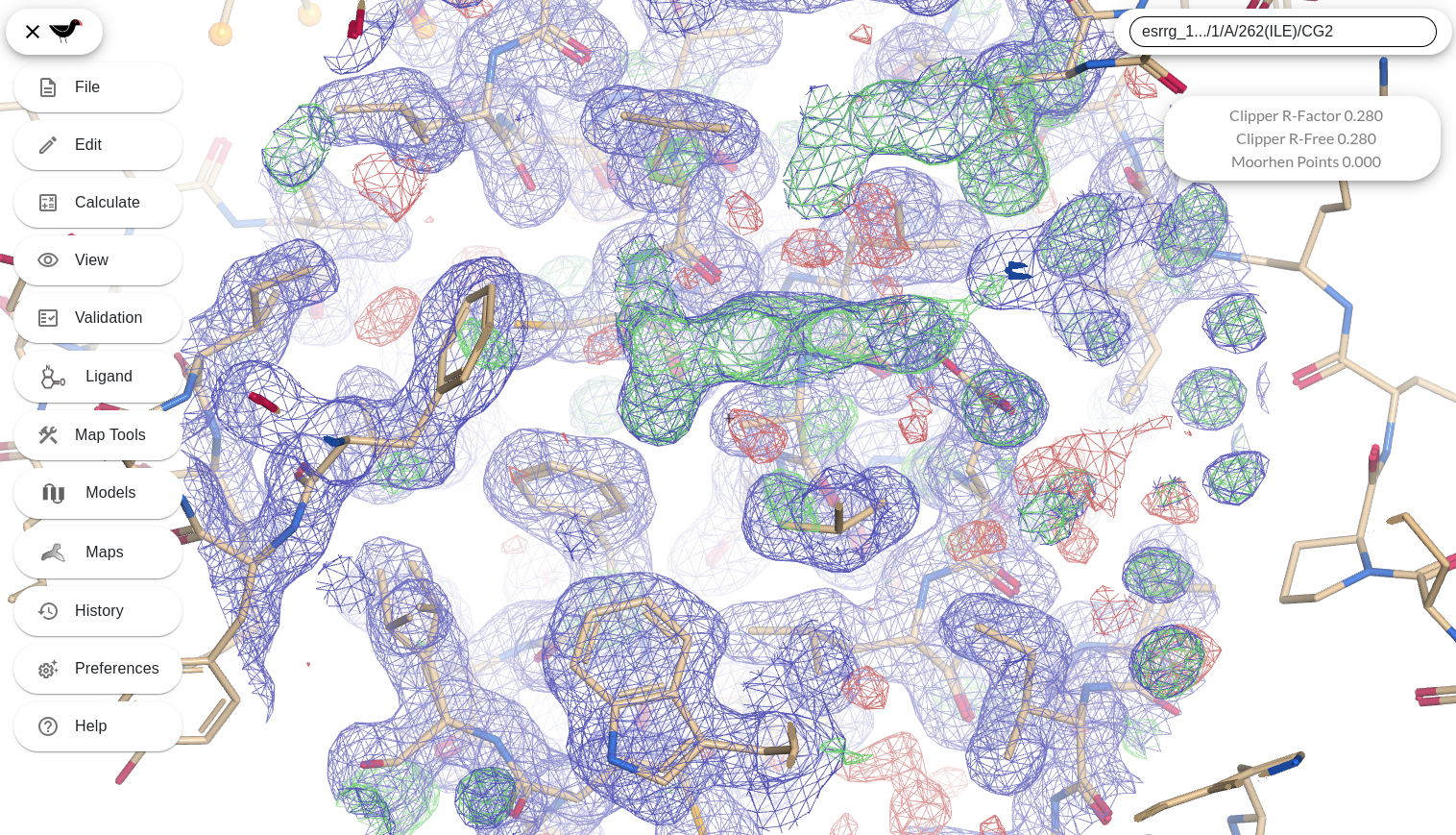
Razglejte se – identificirajte kanale topila in preverite “neskončnost” karte elektronske gostote.
Primerjajte običajno (2mFo–Fc, modre barve) ter diferečno (mFo-Fc, zelene in rdeče barve) karto elektronske gostote. Kaj nam kažeta?
Pozor! Ko enkrat naložite podatke pazite, da ne osvežite strani ali zaprete brskalnika!
Hitri tečaj za Moorhen
vrtenje pogleda: pritisnite in držite levi miškin gumb ter vlevite v levo/desno/bavzgor/navzdol
premikanje: pritisnite in držite sredinski miškin gumb ter vlevite v levo/desno/bavzgor/navzdol
centriranje pogleda na nek atom: kliknite na atom s sredinskim miškinim gumbom
odprite hitri meni za piljenje: kliknite na desni miškin gumn
približevanje/oddaljevanje pogleda: miškin kolešček
sprememba nivoja kontutiranja karte elektronske gostote: CTRL + miškin kolešček (pazite, katera karta je izbrana kot aktivna)
Gradnja in piljenje modela strukture
Model strukture ESRRG je skoraj dokončno zgrajen, kljub temu pa prisotnih nekaj napak in pomankljivosti, med drugim:
nekateri deli so popolnoma napačno postavljeni (niso v skladu z eksperimentalnimi podatki),
nekateri ostanki imajo dihedralne kote glavne verige izven dovoljenih območij,
končni del verige ni zgrajen,
nekateri ostanki bi morali imeti alternativno konformacijo,
manjkajo ligandi,
manjkajo molekule vode.
Nekaj namigov, kaj popraviti:
Med gradnjo opazujte spreminjanje diferenčne karte elektronske gostote ter vrednosti faktorja R.
Namigi za gradnjo:
Izberite drug rotamer, nato pa naredite še piljenje v realnem prostoru (real space refinement), včasih zadostuje zgolj piljenje v realnem prostoru.
Stransko skupino je potrebno obrniti za 180°. Kaj s tem dosežemo v smislu interakcij s sosednjimi skupinami?
Popraviti je potrebno položaj daljšega odseka, izberite vsaj regijo V325–A327. Za izbiro regije držite SHIFT in enkrat kliknite na katerikoli atom prvega ostanka v željenem odseku, nato pa še na katerikoli atom zadnjega. Popravek naredite tako, da najprej malce ročno povlečete regijo v elektronsko gostoto, na koncu pa naredite še piljenje v realnem prostoru.
Ta ostanek ima alternativno konformacijo. Dodajte jo, zanjo izberite drug konformer, ter zaključite s piljenjem v realnem prostoru.
Seveda, preverite aminokislinsko zaporedje kristaliziranega konstrukta in jih dodajte toliko, kolikor jih lahko glede na elektronsko gostoto ter jih lepo umestite vanjo. Lahko zgradite popolnoma ves C-konec?
Analiza oligomernega stanja
Transkripcijsko aktivna oblika ESRRG je dimer.
Koliko verig je v asimetrični enoti?
So med verigami iz sosednjih asimetričnih enot obsežnejši kontakti, ki bi lahko kazali na to, da bi omogočali stabilno oligomerno stanje v raztopini? (Namig: uporabite strežnik PDBePISA.)
Podatki pri nižji ločljivosti
Oglejte si še, kako izgleda elektronska gostota pri nižji ločljivosti in sicer pri 3,2 Å. Gre za strukturo mišjega ESRRG v kompleksu z neko drugo spojino.
Pobrišite vse, kar imate naloženo v Moorhen: Menu > File > Delete everything, ali preprosto ponovno naložite spletno stran.
Naložite v zbirki deponiran model strukture in že izračunane karte elektronske gostote: Menu > File > Fetch from online services (PDBe) > 1VJB (vključite fetch data for map) > Fetch
Oglejte si elektronsko gostoto in razmislite, kako je graditi model strukture pri taki ločljivosti…