S12#
Avtor: Karin Kunstelj
Datum izdelave: 2024-05-25
Koda seminarja: S12
Vhodni podatek#
Povezava do datoteke z vhodnim podatkom: S12
Rezultati analiz#
Ime, velikost in izvorni organizem#
Ime: živalska hem peroksidaza (GenBank: CUR54590.1)
Velikost: 524 aminokislinskih ostankov
Izvorni organizem: neznana bakterija, okoljski vzorec
Identifikacija#
Identifikacija je bila narejena z uporabo NCBI BLAST, varianta blastx (zbirka env_nr). Dobila sem zadetek s 100 % identičnostjoin E vrednostjo O, zato sklepam, da predstavlja celoten zapis za moj protein. Gre torej za živalsko hem peroksidazo iz okoljskega vzorca in neznane bakterije.
>CUR54590.1 Animal heme peroxidase [metagenome] MGVSGQSEPRHGRDFFHIEGEGFVGDPPGTQRLAEEPPPFRFSRMGPRGRRLSRSLRVKLARAMTSGTNR NGTIPAGFTYLGQFVDHDLTFDFTDVALGDDVSPADLLQGRSPTLDLDNVYGAGPNDTKSARFYEDGLHL KIGTTTRAGSDRAKVGFDVPRVGTGGVVARRRAQIPDRRNDENLAVAQTHAAMIRFHNRVVDKQRDTVPP SDRFRRARKRVVLHYQWMLRTDYLPRICDPAVVDDVFTNGRKVFEVGADPLSMPTMPVEFSVAAFRLGHS MVREAYDWNARFPSGQGLLDLLFTFSATSGDLGFNDTLPSNWIADWRRLYRFHEIQRDDLKPPPKEFNFA RRLDTVLVDPLSVLPPGSFGGADEDAGTIEANLAFRNLTRATMLKLASGQQMADLMIAAGVSLTKLTAAQ IRTGLDGTTLEDLTASEREVFVANTPLWFYVLREAELNNGRLTGVGARIVAETFHRAMEGSRFSIVRETD FSPKLGAVPGRFTMTDLLVFAFENKVGLLAPLGD
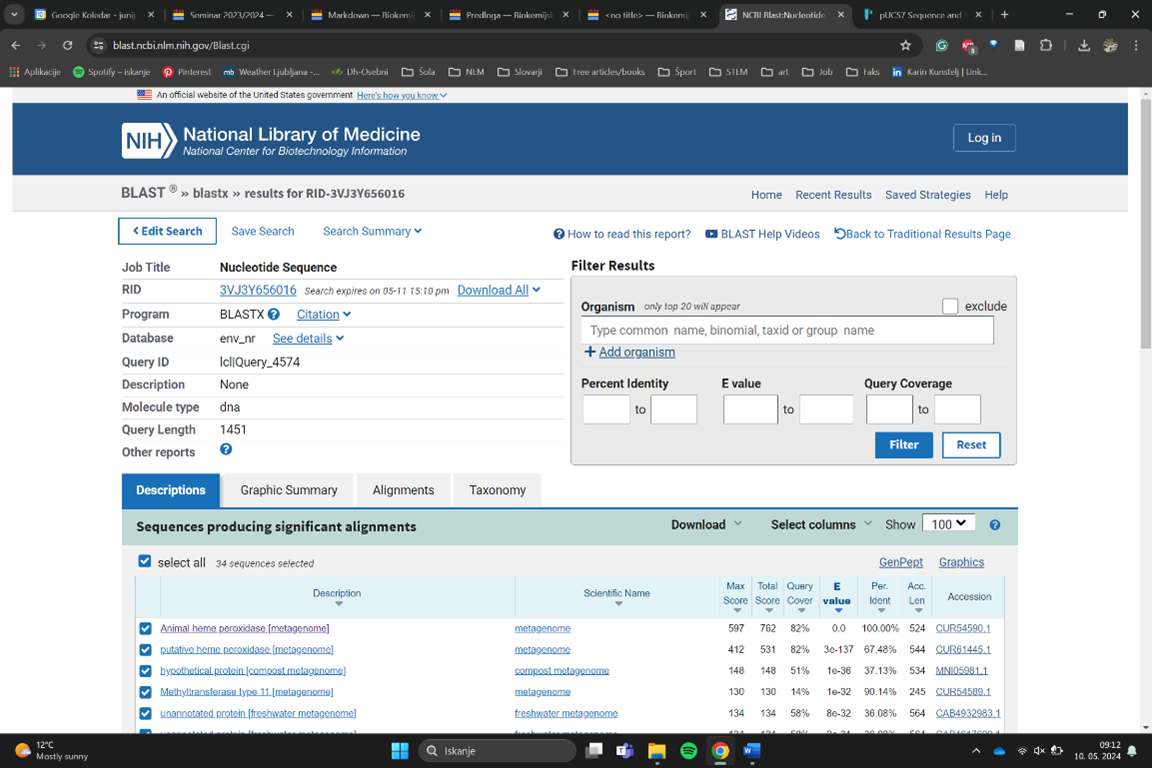
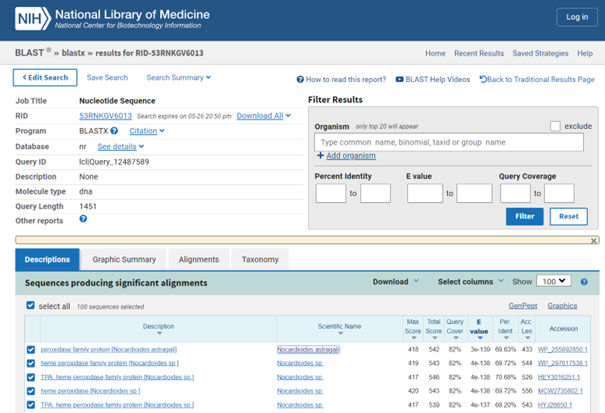
V kolikor pa vzamemo za indentifikacijo v blastx zbirko nr, se pojavijo slabši rezultati z identičnostjo pod 74% in E vrednostjo pod 3e -139. Pri rezultatih z najvišjim ujemanjem v tej zbirki gre v vsakem primeru za protein iz družine hem peroksidaz, organizem pa naj bi bil rodu Nocardioides.
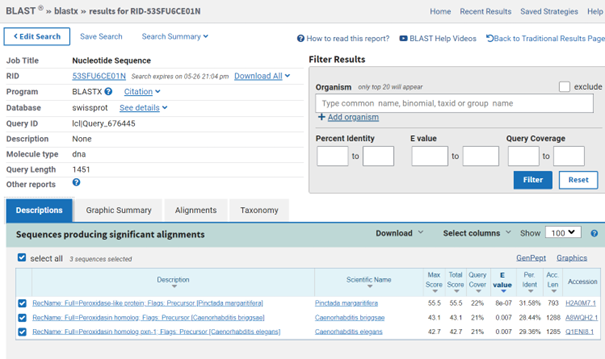
Anotirani rezultati, ki jih dobimo z iskanjem po blastx (zbirka swissprot), pa imajo E vrednost pod 8e -7 in identičnost pod 32%. Gre za protein podoben peroksidazi.
Podobnost z evkariontskimi proteini#
Ker gre za ŽIVALSKO peroksidazo bi seveda pričakovali dobro ujemanje z ekvariontskimi proteini, a temu ni tako.
Post translacijske modifikacije, domene, lokacija in interakcija z drugimi proteini#
Anotirani protein ima v UniProt žal zapisano samo, kateri del zaporedja je signalno zaporedje, tega pa pri ostalih ni. Lokacija naj bi bila za anotirani protein določena po podobnosti -nahaja se izven celice oz. je izločen- , to pa se že preveč oddaljeno našemu proteinu, da bi lahko res zatrdili točno nahajanje. Domene niso nikjer zapisane, prav tako pa niso nikjer omenjene interakcije z drugimi proteini.
Funkcija in vezava substrata#
Ker gre za hem peroksidazo, lahko sklepamo, da gre za encim, ki uporablja vodikov peroksid kot akceptor elektronov. V reakcijo je vključeno železo.
Model proteina#
Z nova različico programa AlphaFold -AlphaFold3- sem dobila izrisan model strukture proteina, ki ima izjemno visok plDDT za večino strukture, torej lahko skoraj z zagotovostjo vidimo, kako približno bi izgledala struktura živalske hem peroksidaze.
