VAJA: Grafi in program Cytoscape#
Pri vaji si bomo na primeru biološke mreže (uporabili bomo mrežo medproteinskih interakcij, protein-protein interaction network, PPIN) pogledali, kako lahko na tak sistem apliciramo teorijo grafov in si z njo pomagamo pri analizi večje količine podatkov. Uporabljali bomo prosto dostopen program Cytoscape.
Program Cytoscape že sam po sebi omogoča nekatere analize, dodatne analize, uvoz podatkov iz nekaterih zbirk ipd. pa omogočajo vtičniki oz. aplikacije (Apps). Tudi mi jih bomo nekaj uporabili, zato jih bomo najprej namestili oz. preverili, ali so že nameščeni; če so, bomo preverili, ali jih je morda potrebno nadgraditi. Do namestitve vtičnikov dostopamo preko glavnega medija Apps/App Manager…. Potrebovali bomo naslednje aplikacije:
STRING, ki omogoča uvoz podatkov o interakcijah protein-protein in protein-majhne molekule iz različnih zbirk ter njihovo funkcijsko obogatitev s podatki GO (Gene Ontology);
ClusterViz, ki v mreži z uporabo različnih algoritmov identificira skupke;
BiNGO, ki točkam v mreži pripiše termine GO (Gene Ontology) ter analizira, katere kategorije so statistično bolj zastopane kot druge - na tak način lahko recimo ugotovimo, katere točke oziroma podmreže so vključene v določen biološki proces, ko analiziramo mreže genov ali proteinov.
Naloga#
V interaktomu (mreža medproteinskih interakcij) človeka ali katerega drugega organizma identificirajte skupke in poglejte, ali proteini v skupkih opravljajo sorodno funkcijo oz. so vključeno v isti osnovni biološki proces.
Postopek#
V Cytoscape uvozite celoten interaktom izbranega organizma. Pazite - v primeru, ko je interaktom velik, lahko uvažanje podatkov traja precej časa!
V oknu, do katerega pridete preko menija File/Import/Network/Public Databases…, izberite kot Data Source “Universal Interaction Database Client”, pod Search Mode pa izberite “Import Interactome”, nato pa lahko izberete vrsto oziroma organizem. Opomba: če želite prenesti interakcije, ki vključujejo samo določen protein oz. gen, lahko iskanje opravite preko “Search by ID (gene/protein/compound ID)”. Pritisnite gumb Search.
V polju spodaj se vam prikažejo posamezne zbirke interakcij, ki jih lahko uvozite. Izberite samo eno, ki ni prevelika (recimo, naj vsebuje manj kot 10 000 zapisov oz. interakcij). Interaktom lahko načeloma uvozimo iz večih zbirk hkrati za isti organizem, nato pa mreže zlijemo (funkcija Automatic Network Merge, lahko pa združevanje opravimo naknadno). V razmislek: na osnovi česa bi lahko mreže med seboj zlivali?
Kliknite na Import, počakajte, da se podatki prenesejo s spleta, in v oknu, ki se za tem odpre, kliknite na Close. Združevanje zdaj ni potrebno, saj smo uvozili samo eno mrežo. Zaprimo tudi okno za uvoz interaktoma. Tako nam ostane osnovno okno Cytoscape, ki vsebuje uvožene podatke.
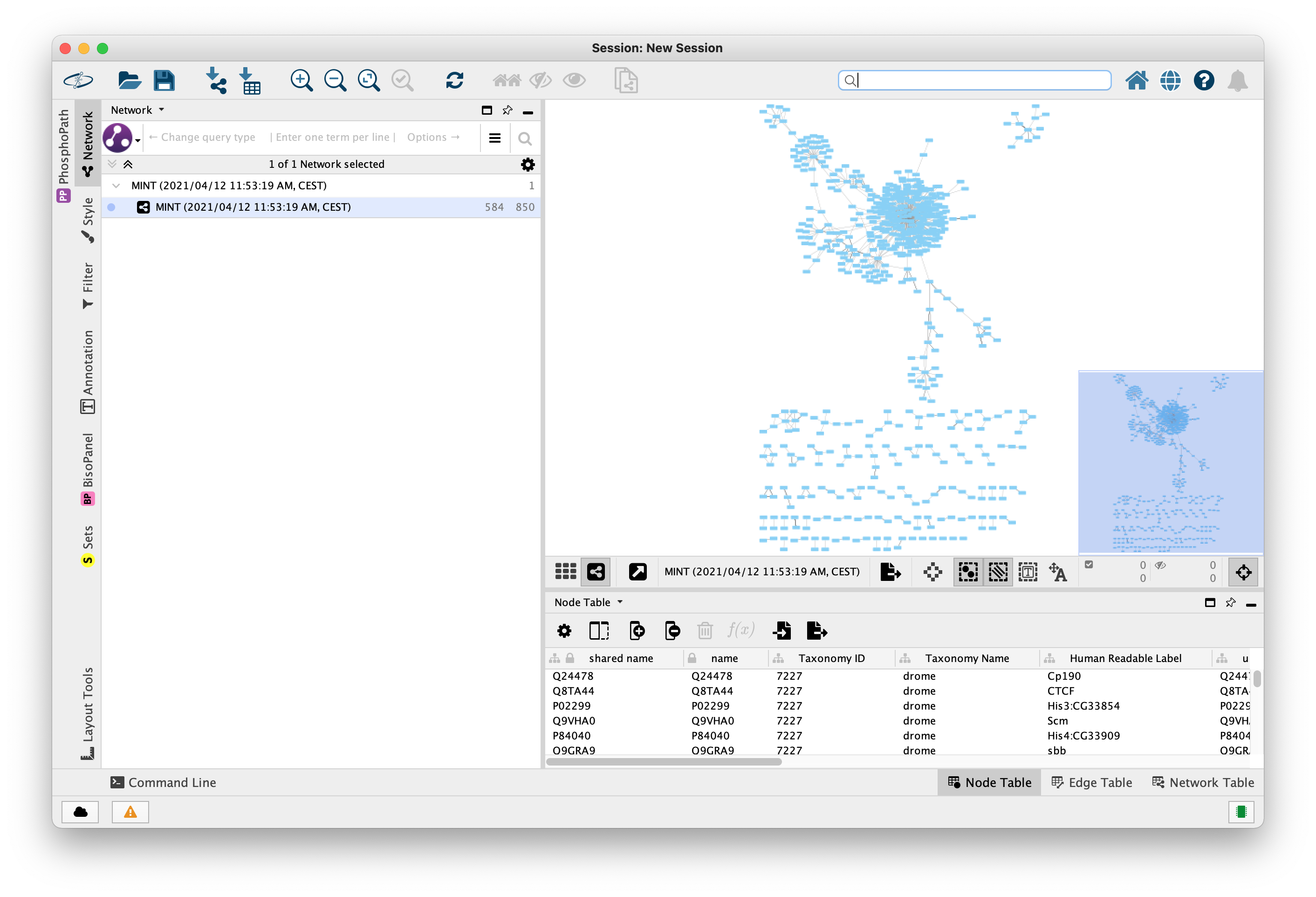 Uvoz interaktoma iz zbirke MINT za organizem Drosophila melanogaster.
Uvoz interaktoma iz zbirke MINT za organizem Drosophila melanogaster.
Privzet prikaz je Perfuse Force Directed Layout. Prikaz lahko spreminjate preko menija Layout. Algoritmi in z njimi povezanimi načini razporeditve točk so podrobneje opisani v ustreznem razdelku navodil za program Cytoscape - Automatic Layout Algorithms.
Mrežo lahko približate in se premikate po njej s pomočjo miške (kolešček za približevanje/oddaljevanje, levi gumb za premikanje), lahko tudi izbirate posamezne vozle in povezave (levi gumb), za približevanje/oddaljevanje lahko uporabite tudi gumb s simbolom lupe v meniju. Točke lahko tudi premikate; na izhoden način organizacije vozlov se lahko vrnete preko menija Layout in izbiro ustreznega načina vizualizacije.
Mreže, ki ste jih uvozili iz nekaterih zbirk, lahko vsebujejo vzporedne povezave ali pa samo-povezave (zanke na isto točko); le-te lahko odstranite s funkcijo Edit/Remove Duplicated Edges oz. Edit/Remove Self-Loops.
Oglejte si tabele pod grafom - Node Table - tabela točk, Edge Table - tabela povezav, Network Table - tabela mrež. Kaj vsebujeta tabeli točk in povezav?
Analizirajte mrežo (določite nekatere osnovne parametre mrež, ki so bili predstavljeni na predavanjih - poglejte slike s predavanj!) preko menija Tools/NetworkAnalyzer/Network Analysis/Analyze Network. Izberite, da je mreža neusmerjena (undirected). Rezultati se prikažejo v posebnem oknu Results Panel (pazite, lahko je skrito kje v ozadju). Na primer, nekateri osnovni parametri so prikazani kot številčne vrednosti, nekateri pa v obliki diagramov, na primer porazdelitev stopnje točk; pri slednji je y-os logaritemska (pod Chart Settings v oknu z rezultati lahko spremenite, ali bosta osi logaritemski ali ne). Lahko dodate “Power Law”, ki vam na podatke prilega porazdelitev, značilno za scale-free mreže. Kakšna je vaša mreža? Je to v skladu s pričakovanji in zakaj da/ne?
Po analizi se izračunani parametri avtomatsko dodajo tudi v tabelo točk, kjer lahko točke razvrščate glede na vrednosti teh parametrov. Oglejte si točke, ki imajo najvišje/najnižje vrednosti in povežite to z njihovo umestitvijo v graf (število sosedov oz. njihova stopnja in podobno).
Stil (izgled točk in povezav, tudi glede na parametre v tabeli) lahko prilagajate tudi preko polja Control Panel, kjer namesto Network izberite Style, na dnu pa potem izbirate med Node, Edge ali Network. Sprememba stila deluje na izbrane točke/povezave; če ga želite aplicirati na celotno mrežo, ne izberite nobene točke ali pa izberite vse točke.
Če bi želeli celico tega organizma, na katerem delate analize, uničiti… Katere proteine oz. katere gene bi ciljali?
Sprememba stila, ki ga lahko aplicirate na točke in/ali povezave.
Preko menija Apps/ClusterViz/Open Viz zaženite aplikacijo ClusterViz in izberite način MCODE. Poiščite skupke s privzetimi nastavitvami, nato pa nastavitve spremenite in poglejte, kako se spremenijo rezultati; le-ti so prikazani v posebnem polju, ki ga lahko odpnete z glavnega okna programa Cytoscape. Ko izberete nek skupek, se točke tega skupka označijo/izberejo v celotni mreži (rumena barva). Opomba: med rezultati iskanja skupkov in drugimi rezultati lahko preklapljate s puščicami v Results Panel). Posamezen skupek lahko tudi skopirate v pod-mrežo (Create Sub-Network).
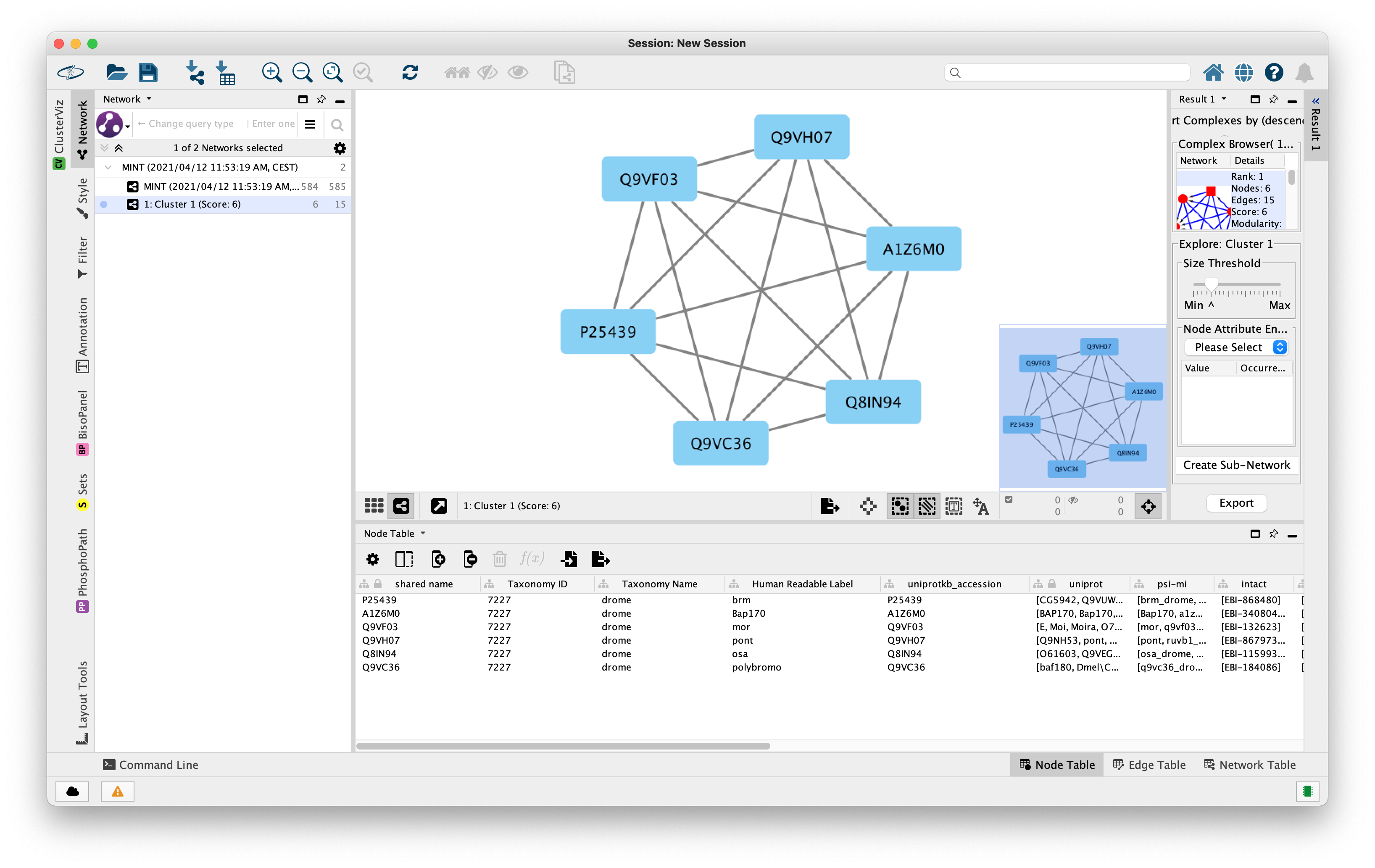 En skupek iz interaktoma organizma Drosophila melanogaster, ki je pravzaprav popoln graf.
En skupek iz interaktoma organizma Drosophila melanogaster, ki je pravzaprav popoln graf.
Poizkusite ugotoviti, ali identificirani skupki ustrezajo določeni funkciji. To lahko naredite s pomočjo programa BiNGO, ki identificira statistično gledano bolj pogoste termine GO (Gene Ontology); aplicirate ga lahko na celotno mrežo, mi pa ga bomo uporabili na posameznih skupkih.
Zaženite program preko menija Apps/BiNGO.
Izberite skupek, ki ga želite analizirati (rezultati analize z ClusterViz - to okno si pustite odprto!).
V oknu programa BiNGO izberite možnost “Get Cluster from Network”. Vsak skupek (cluster) morate poimenovati z unikatnim imenom (če tega ne storute vas bo program sam opozoril). Preglejte parametre, preverite izbrani organizem. Analize lahko gelate glede na biološki proces, funkcijo, celično lokalizacijo, … (te možnosti so pod Select ontology file). Zaženite analizo.
Rezultati analize so prikazani kot tabela (urejena glede na vrednost p obogogatitve posameznega termina) in pa kot mreža terminov; v slednji lahko prikaz prilagodite na izračunane parametre (recimo vrednost p).
Za tak tip analize je primerna hierarhična vizualizacija - oglejte si jo (Layout/Hiearchial Layout).
