S02#
Avtor: Lara Ferjančič
Datum izdelave: 2024-05-10
Koda seminarja: S02
Vhodni podatek#
Povezava do datoteke z vhodnim podatkom: S02
Rezultati analiz#
Sekvenca za protein v vhodni sekvenci#
Celotna vhodna sekvenca ni bila samo zapis za protein, ampak je vsebovala tudi dele plazmida pUC57, v katerega je bila sekvenca za protein vstavljena. Vstavljen zapis je bil določen preko globalne poravnave Needleman-Wunsch - 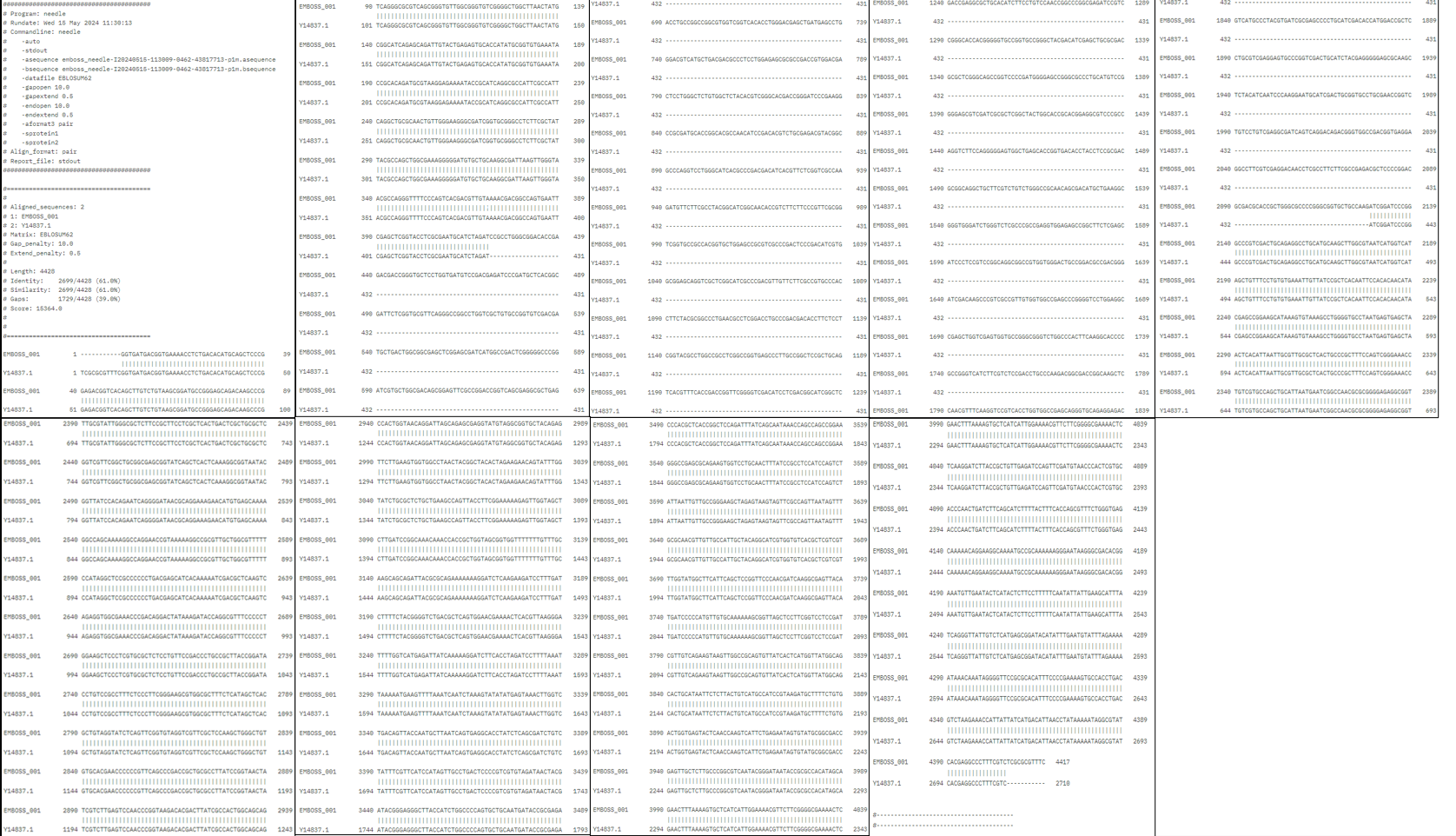 celotne sekvence plazmida pUC57 in vhodne sekvence kaže, kateri del je vstavljen v plazmid. Del, kjer se sekvenci razlikujetai, predstavlja vstavljen zapis.
celotne sekvence plazmida pUC57 in vhodne sekvence kaže, kateri del je vstavljen v plazmid. Del, kjer se sekvenci razlikujetai, predstavlja vstavljen zapis.
Ime proteina in njegov izvorni organizem#
Sledila je identifikacija proteina z orodjem blastx, kjer je bila kot baza podatkov uporabljena baza metagenomskih proteinov (Metagenomic proteins). Ta baza je bila uporabljena, saj z uporabo drugih baz ni bilo do 100 % ujemanja v sekvenci pri zadetkih. Z uporabo baze podatkov metagenomskih proteinov je bila možna identifikacija proteina - prvi zadetek iskanja je imel 100 % ujemanje (Percent Identity), iz česar lahko sklepamo, da vhodna sekvenca vsebuje zapis za protein 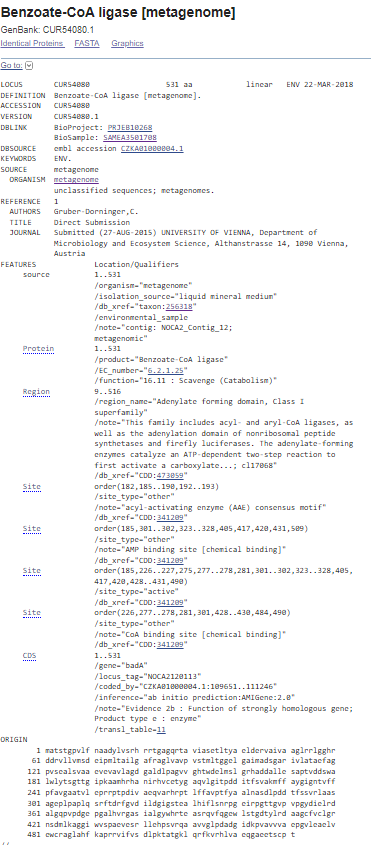 . Rezultati tega iskanja so prikazani na
. Rezultati tega iskanja so prikazani na 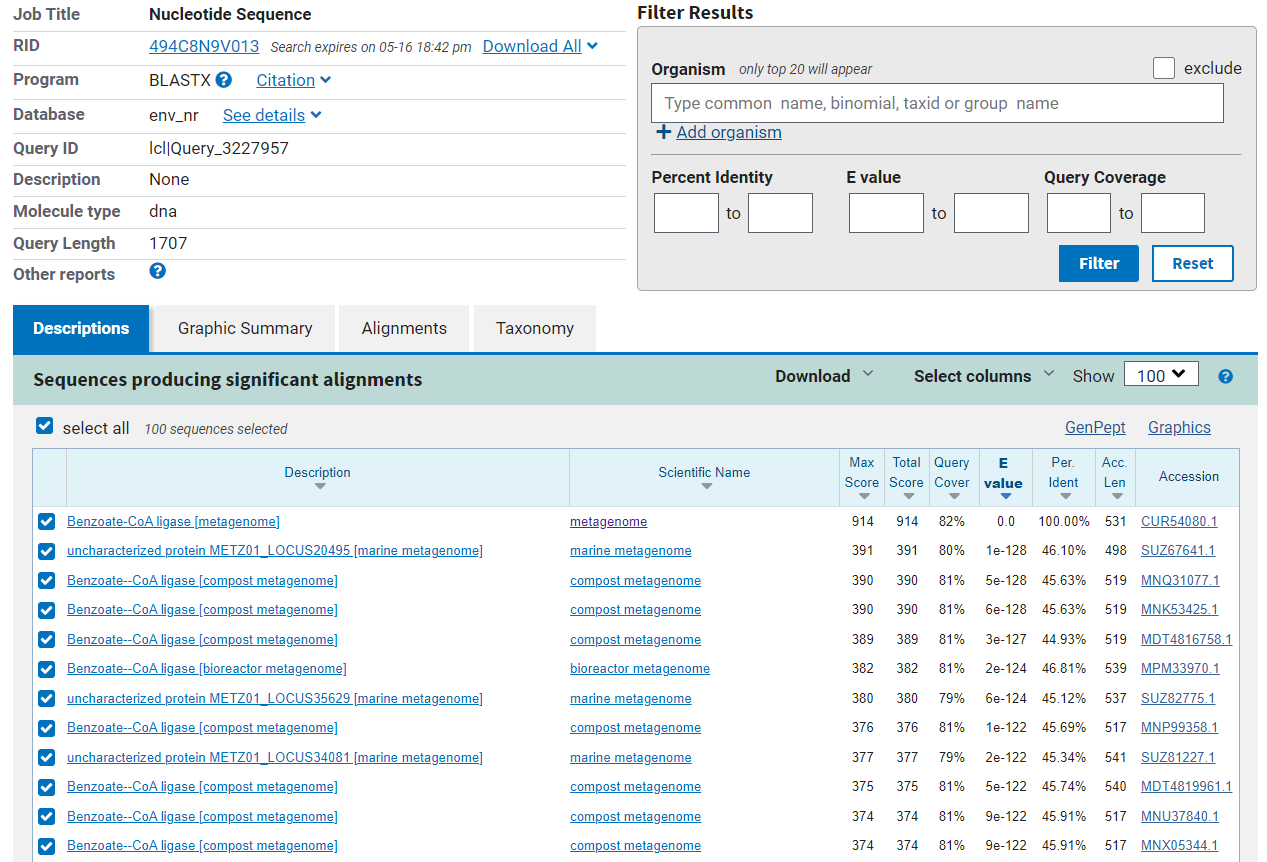 .
V plazmid ni bila vstavljena celotna sekvenca za ta protein, na kar kaže delež proteina, ki se ujema z uporabljeno sekvenco (Query Cover) pri tem iskanju, ki znaša 82 %.
.
V plazmid ni bila vstavljena celotna sekvenca za ta protein, na kar kaže delež proteina, ki se ujema z uporabljeno sekvenco (Query Cover) pri tem iskanju, ki znaša 82 %.
Točen izvorni organizem tega proteina ni poznan, znano je, da gre za sev A2 bakterijska reda Nocardioides. Poznan je tudi 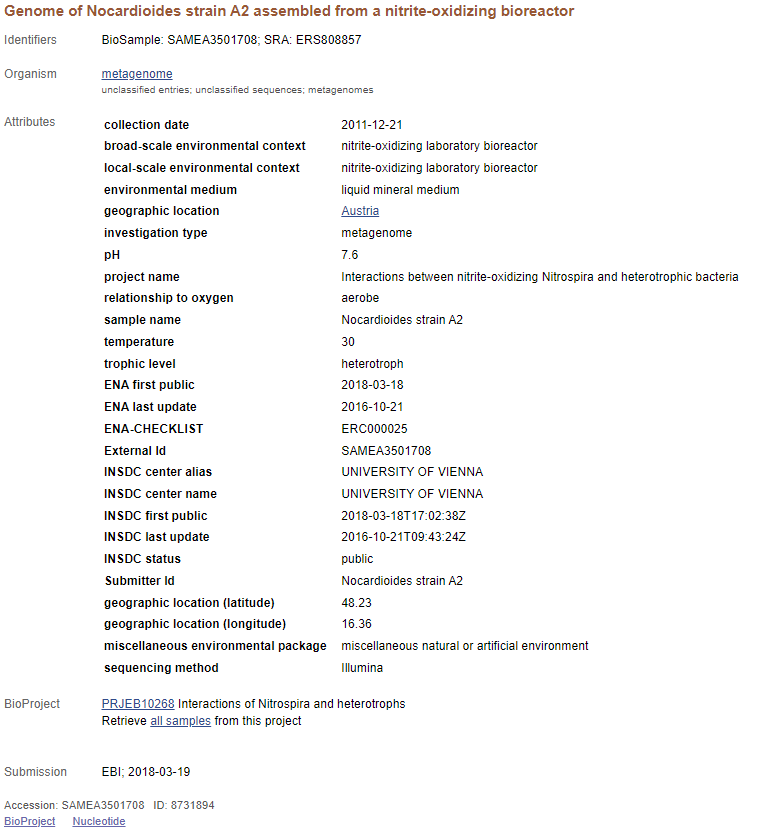 , iz katerega so pridobili sekvenco - vzorec izhaja iz nitrit-oksidirajočega laboratorijskega bioreaktorja, v katerem je bila temperatura 30 °C in pH 7,6.
, iz katerega so pridobili sekvenco - vzorec izhaja iz nitrit-oksidirajočega laboratorijskega bioreaktorja, v katerem je bila temperatura 30 °C in pH 7,6. 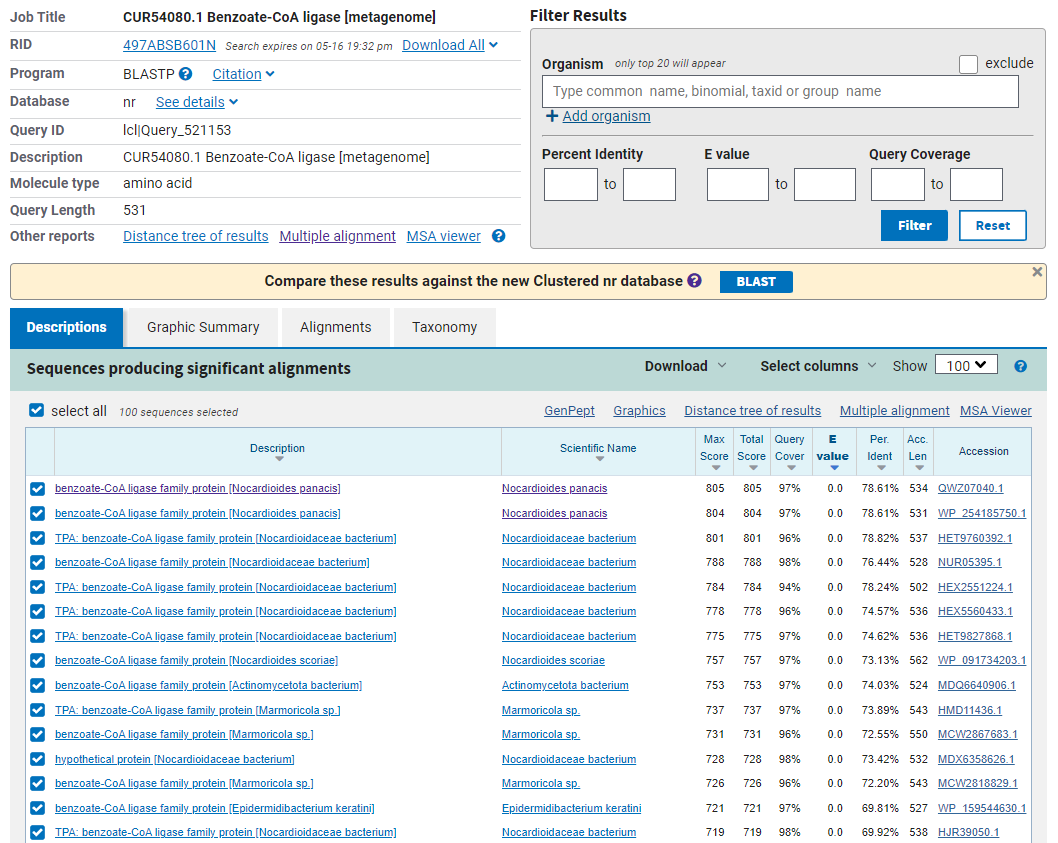 izvirajo iz organizma sorodnih taksonomskih stopenj, kar kaže, da gre res za protein iz proteina reda Nocardioides.
izvirajo iz organizma sorodnih taksonomskih stopenj, kar kaže, da gre res za protein iz proteina reda Nocardioides.
Lokalizacija in velikost proteina#
Protein sestavlja 531 aminokislinskih ostankov. Molekulska masa proteina znaša 56,3 kDa, izračun z orodjem  .
Celična lokacija za ta protein in njemu podobne proteine, ni poznana. Iskanje podobnih proteinov je poteklo z orodjem blastp, izbrane so bile
.
Celična lokacija za ta protein in njemu podobne proteine, ni poznana. Iskanje podobnih proteinov je poteklo z orodjem blastp, izbrane so bile  (non-redundant) in nato še iskanje po
(non-redundant) in nato še iskanje po 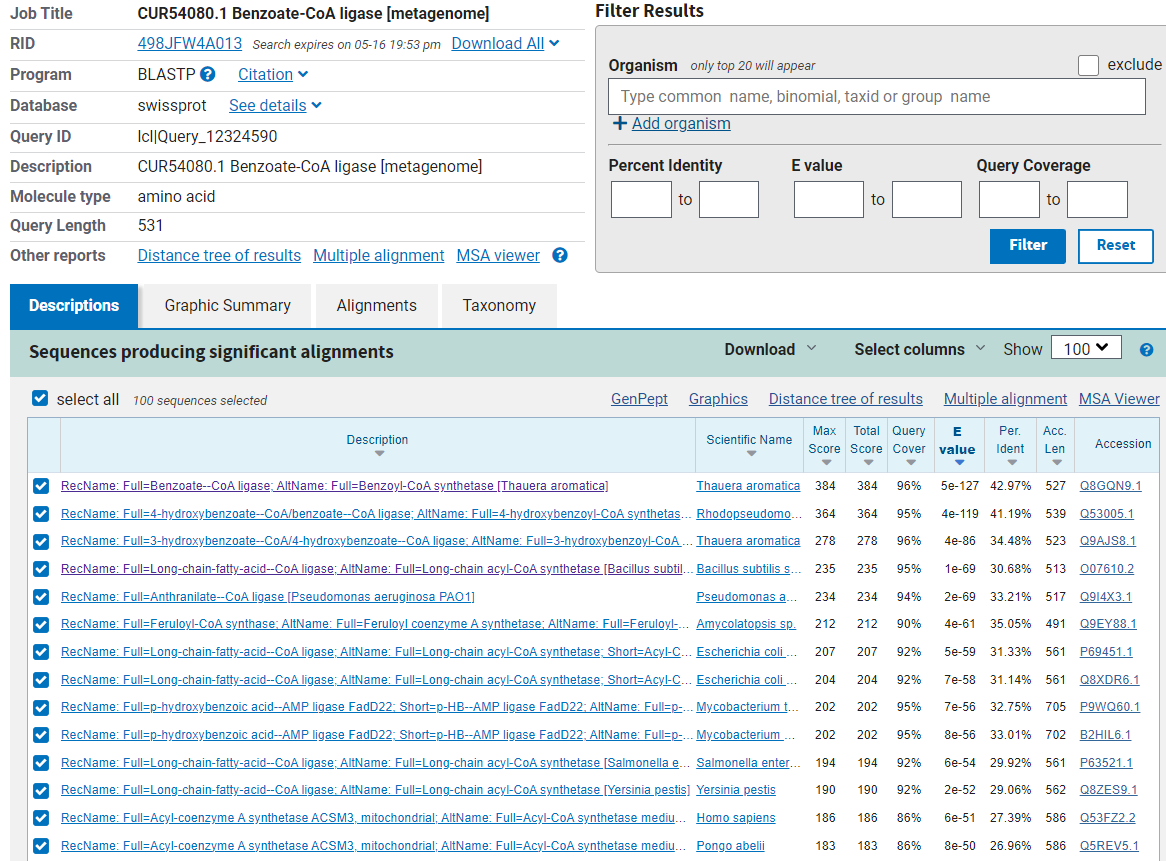 . Glede na funkcijo proteina in strukturo podobnih proteinov, ki ne vsebujejo transmembranskih regij, lahko sklepamo, da se protein nahaja v citoplazmi bakterijskih celic. Prav tako AlphaFold3 model protein napove kot globularni protein in ne kot protein s transmembransko regijo, iz česar lahko sklepamo, da ne gre za membranski protein. Pravtako encim spada med ligaze, ki se najpogosteje nahajajo v citoplazmi, kar še dodatno podkrepi predvidevanje, da se nahaja v citoplazmi. Za določitev, kje se protein dejansko nahaja v celici, bi bila najbolj zanesljiva eksperimentalna določitev.
. Glede na funkcijo proteina in strukturo podobnih proteinov, ki ne vsebujejo transmembranskih regij, lahko sklepamo, da se protein nahaja v citoplazmi bakterijskih celic. Prav tako AlphaFold3 model protein napove kot globularni protein in ne kot protein s transmembransko regijo, iz česar lahko sklepamo, da ne gre za membranski protein. Pravtako encim spada med ligaze, ki se najpogosteje nahajajo v citoplazmi, kar še dodatno podkrepi predvidevanje, da se nahaja v citoplazmi. Za določitev, kje se protein dejansko nahaja v celici, bi bila najbolj zanesljiva eksperimentalna določitev.
Domenska zgradba#
Določitev domenske zgradbe je potekla s programom 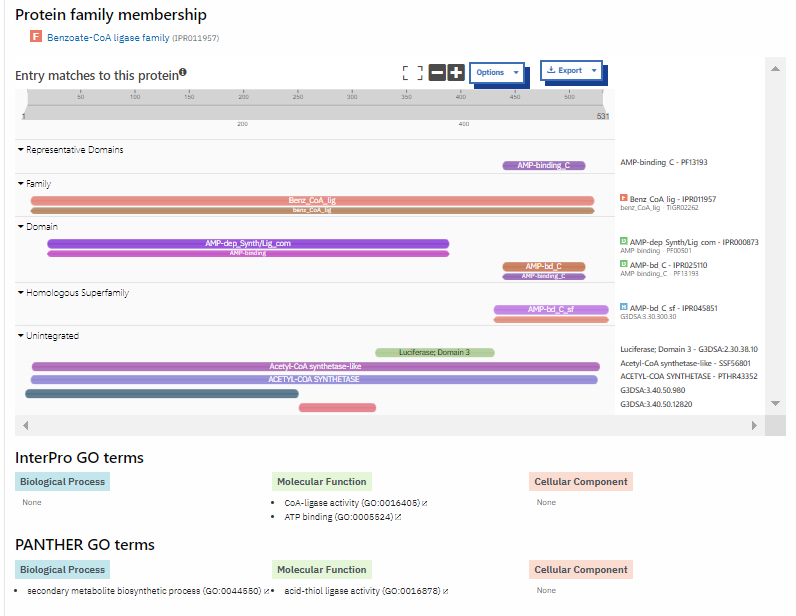 . Rezultati, ki so zapisani v razdelku domene (Domain), kažejo, da ima ta protein od AMP odvisno ligazno oziroma sintetazno domeno ali pa gre za AMP vezavni encim. Rezultati tega iskanja se ujemajo z poznanimi informacijami o temu proteinu, kot je ime in funkcija proteina.
Hkrati pa rezultati v istem razdelku kažejo na to, da ima protein tudi AMP vezavno domeno na C koncu proteina. Ta domena najverjetnje regulira aktivnost od AMP odvisne ligazne oziroma sintetazne domene. Aminokislinski ostanki 25-386 sestavljajo od AMP odvisno ligazno domena, AMP vezavno domeno pa aminokislinski ostanki 436-509.
. Rezultati, ki so zapisani v razdelku domene (Domain), kažejo, da ima ta protein od AMP odvisno ligazno oziroma sintetazno domeno ali pa gre za AMP vezavni encim. Rezultati tega iskanja se ujemajo z poznanimi informacijami o temu proteinu, kot je ime in funkcija proteina.
Hkrati pa rezultati v istem razdelku kažejo na to, da ima protein tudi AMP vezavno domeno na C koncu proteina. Ta domena najverjetnje regulira aktivnost od AMP odvisne ligazne oziroma sintetazne domene. Aminokislinski ostanki 25-386 sestavljajo od AMP odvisno ligazno domena, AMP vezavno domeno pa aminokislinski ostanki 436-509.
Rezultati iskanja kažejo, da se del aminokislinskega zaporedja ujema z zaporedjem luciferzane domene 3. Tudi v drugih iskanjih (iskanje podobnih proteinov z blast, zadetki z bisteveno nižjo podobnostjo) se med zadetki nahajo različne luciferaze. Glede na to da funkcija obeh proteinov ni podobna, ampak se ujemata samo v delu zaporedja, obstaja možnost, da je prišlo do duplikacije tega dela zaporedja v genomu tekom evolucije.
Post-translacijske modifikacije#
Za preiskovani protein ni poznanih post-translacijskih modifikacij. Enako velja za temu proteinu podobne proteine, ki so anotirani v  in za podobne proteine z
in za podobne proteine z 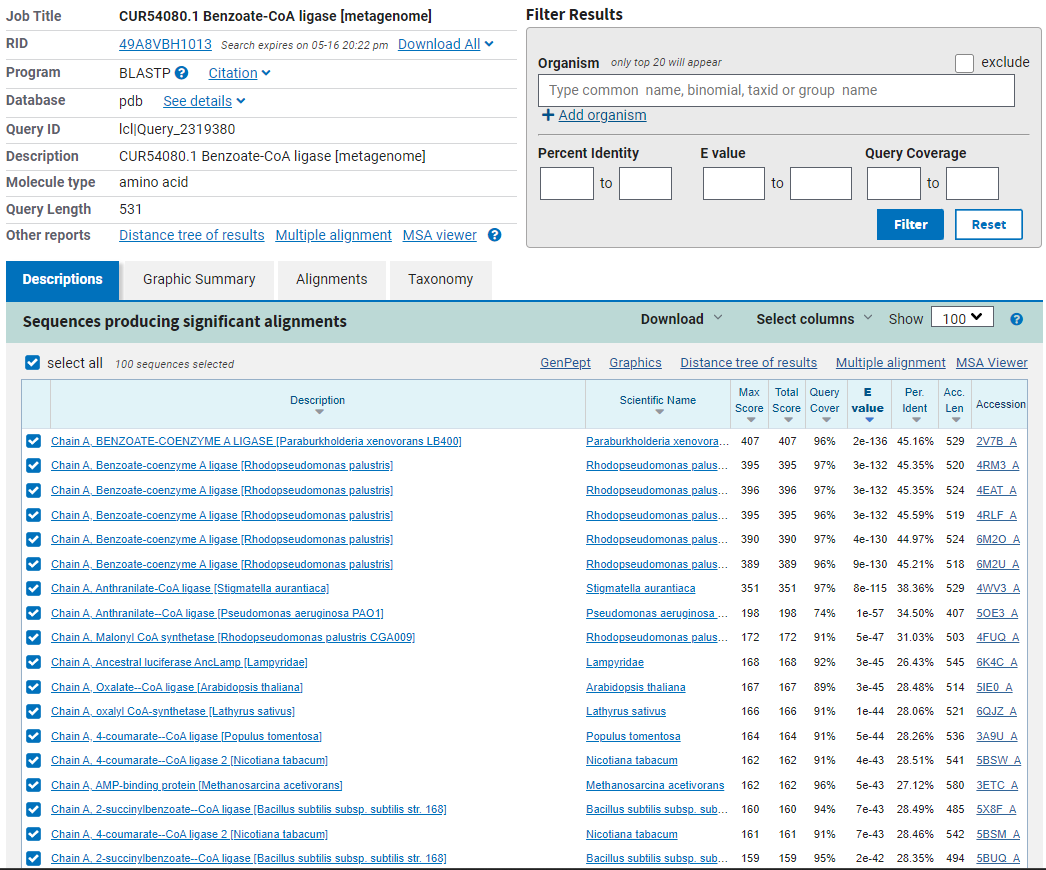 . Glede na to da preko podobnosti s podobnimi proteini ne moremo sklepati na post-translacijske modifikacije, bi bila za določitev post-translacijskih modifikacij tega proteina najboljša možnost določitev le-teh eksperimentalno.
. Glede na to da preko podobnosti s podobnimi proteini ne moremo sklepati na post-translacijske modifikacije, bi bila za določitev post-translacijskih modifikacij tega proteina najboljša možnost določitev le-teh eksperimentalno.
Funkcija proteina#
Funkcije podobnih proteinov, ki jih najdemo z orodjem blastp v bazi podatkov Swiss/UniProt, lahko sklepamo, da je funkcija tega proteina kataliza tvorbe benzoil-CoA iz benzoata in koencima A, ob čimer se porablja ATP. Reakcija je del metabolizma benzoata. Glede na substrate, ki jih lahko uporabijo podobni encimi, lahko sklepamo, da lahko encim namesto benzoata veže tudi benzoatne derivate kot so 4-hidroksibenzoat, 3-hidroksibenzoat in drugi podobni derivati.
 kažejo na enako funkcijo - v zadnjem razdelku rezultatov iskanja vidimo, da preiskovani protein spada v superdružino acetil-CoA sintetazi-podobnih proteinov, ki sintetizirajo kompleks spojine z CoA, ob čimer se porablja ATP. Hkrati ima protein od AMP odvisno ligazno oziroma sintetazno domeno in pa AMP vezavno domeno.
kažejo na enako funkcijo - v zadnjem razdelku rezultatov iskanja vidimo, da preiskovani protein spada v superdružino acetil-CoA sintetazi-podobnih proteinov, ki sintetizirajo kompleks spojine z CoA, ob čimer se porablja ATP. Hkrati ima protein od AMP odvisno ligazno oziroma sintetazno domeno in pa AMP vezavno domeno.
Interakcije z drugimi proteini#
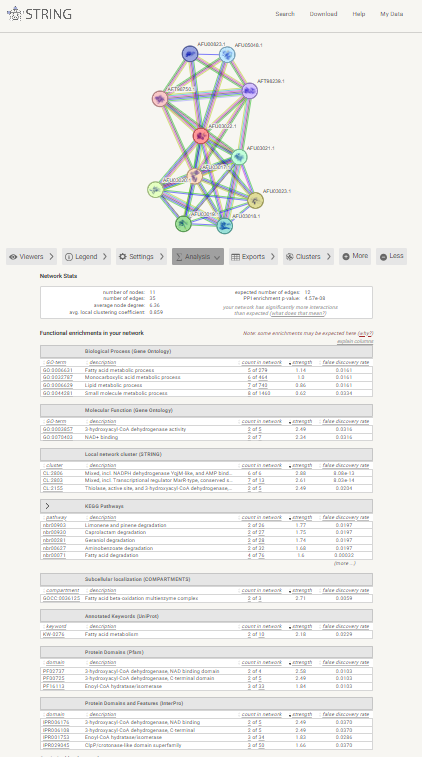 preiskovanega proteina z drugimi proteini je bila opravljena z orodjem String. Ker za preiskovani protein ni točno poznan izvorni organizem, je bila ta analiza opravljena z benzoat-CoA ligazo iz bakterije Nocardia brasiliensis, ki spada v red Nocardioides tako kot preiskovani protein. Hkrati je za ta protein, izmed proteinov, ki bi bili primerni za to analizo, poznanih največ interakcij.
Rezultati analize kažejo, da benzoat-CoA ligaza interagira z osmimi drugimi proteini, rezultati kažejo še na interakcije z dvema hipotetičnima proteinoma. Proteini, s katerimi preiskovan protein interagira, so encimi, ki imajo za substrat koencim A. Ti encimi sodelujejo pri različnih metabolnih procesih, kot so metabolizem lipidov in maščobnih kislin ter metabolizem manjših organskih molekul. Najdene interakcije so bile potrjene eksperimentalno oziroma se preiskovani protein hkrati izraža v celici kot proteini, s katerimi interagira. Interakcije s hipotetičnimi proteini so predvidene iz bližine genov za proteine.
Ker večina teh interakcij ni eksperimentalno potrjenih, bi bilo za natančnejšo analizo interakcij potrebno opraviti ekperimentalno analizo interakcij z drugimi proteini. Hkrati je ob interpretaciji dobljenih rezultatov potrebno upoštevati še dejstvo, da rezultati prikazujejo interakcije benzoat-CoA ligaze iz bakterije Nocardia brasiliensis, ki ni nujno izvorni organizem preiskovanega proteina. Za določitev interakcij preiskovanega proteina bi bilo potrebno najprej identificirati izvorni organizem in potem določiti interakcije točno s tem proteinom in ne s proteinom iz sorodnega organizma.
preiskovanega proteina z drugimi proteini je bila opravljena z orodjem String. Ker za preiskovani protein ni točno poznan izvorni organizem, je bila ta analiza opravljena z benzoat-CoA ligazo iz bakterije Nocardia brasiliensis, ki spada v red Nocardioides tako kot preiskovani protein. Hkrati je za ta protein, izmed proteinov, ki bi bili primerni za to analizo, poznanih največ interakcij.
Rezultati analize kažejo, da benzoat-CoA ligaza interagira z osmimi drugimi proteini, rezultati kažejo še na interakcije z dvema hipotetičnima proteinoma. Proteini, s katerimi preiskovan protein interagira, so encimi, ki imajo za substrat koencim A. Ti encimi sodelujejo pri različnih metabolnih procesih, kot so metabolizem lipidov in maščobnih kislin ter metabolizem manjših organskih molekul. Najdene interakcije so bile potrjene eksperimentalno oziroma se preiskovani protein hkrati izraža v celici kot proteini, s katerimi interagira. Interakcije s hipotetičnimi proteini so predvidene iz bližine genov za proteine.
Ker večina teh interakcij ni eksperimentalno potrjenih, bi bilo za natančnejšo analizo interakcij potrebno opraviti ekperimentalno analizo interakcij z drugimi proteini. Hkrati je ob interpretaciji dobljenih rezultatov potrebno upoštevati še dejstvo, da rezultati prikazujejo interakcije benzoat-CoA ligaze iz bakterije Nocardia brasiliensis, ki ni nujno izvorni organizem preiskovanega proteina. Za določitev interakcij preiskovanega proteina bi bilo potrebno najprej identificirati izvorni organizem in potem določiti interakcije točno s tem proteinom in ne s proteinom iz sorodnega organizma.
Nameravano je bilo opraviti tako analizo še za podobne evkariontske proteine, vendar je bilo to  , saj ni poznanih podatkov interakcij med proteini za najbližje sorodne proteine. Glede na to da imajo evkariontski organizmi vse proteine, s katerimi so potrjene interakcije pri prokariontskih organizmih, najverjetneje evkariontska benzoat-CoA ligaza interagira z evkariontskimi variantani teh proteinov. Najverjetneje pa interagira še z drugimi proteini, kar pa bilo najbolj natančno ugotoviti z eksperimentom.
, saj ni poznanih podatkov interakcij med proteini za najbližje sorodne proteine. Glede na to da imajo evkariontski organizmi vse proteine, s katerimi so potrjene interakcije pri prokariontskih organizmih, najverjetneje evkariontska benzoat-CoA ligaza interagira z evkariontskimi variantani teh proteinov. Najverjetneje pa interagira še z drugimi proteini, kar pa bilo najbolj natančno ugotoviti z eksperimentom.
Sorodni proteini#
Podobne proteine po iskanju z blastp po  je bila izvedena poravnava več zaporedij v
je bila izvedena poravnava več zaporedij v  . Rezultati analize so bili vnešeni v orodje phylo.io in rezultat analize je
. Rezultati analize so bili vnešeni v orodje phylo.io in rezultat analize je  . Drevo prikazuje, da so najbolj sorodni proteini preiskovanemu proteinu druge benzoat-CoA ligaze iz drugih organizmov iz reda Nocardioides in nižjih taksonomskih stopenj tega reda. Tej skupini proteinov so najbolj sorodne benzoat-CoA ligaze iz reda Marmoricola. Glede na to da so v vrhnjem (zgoraj na sliki) delu drevesa najbolj sorodna proteina iz Nocardioides in iz Marmoricola, lahko sklepamo, da je tekom evolucije prišlo večkrat do loćitve posameznih redov.
. Drevo prikazuje, da so najbolj sorodni proteini preiskovanemu proteinu druge benzoat-CoA ligaze iz drugih organizmov iz reda Nocardioides in nižjih taksonomskih stopenj tega reda. Tej skupini proteinov so najbolj sorodne benzoat-CoA ligaze iz reda Marmoricola. Glede na to da so v vrhnjem (zgoraj na sliki) delu drevesa najbolj sorodna proteina iz Nocardioides in iz Marmoricola, lahko sklepamo, da je tekom evolucije prišlo večkrat do loćitve posameznih redov.
Enako analizo lahko naredimo glede sorodnosti preiskovanega proteina s sorodnimi evkariontskimi proteini. Najbolj soroden protein izhaja iz globokomorske spužve Geodia barretti. 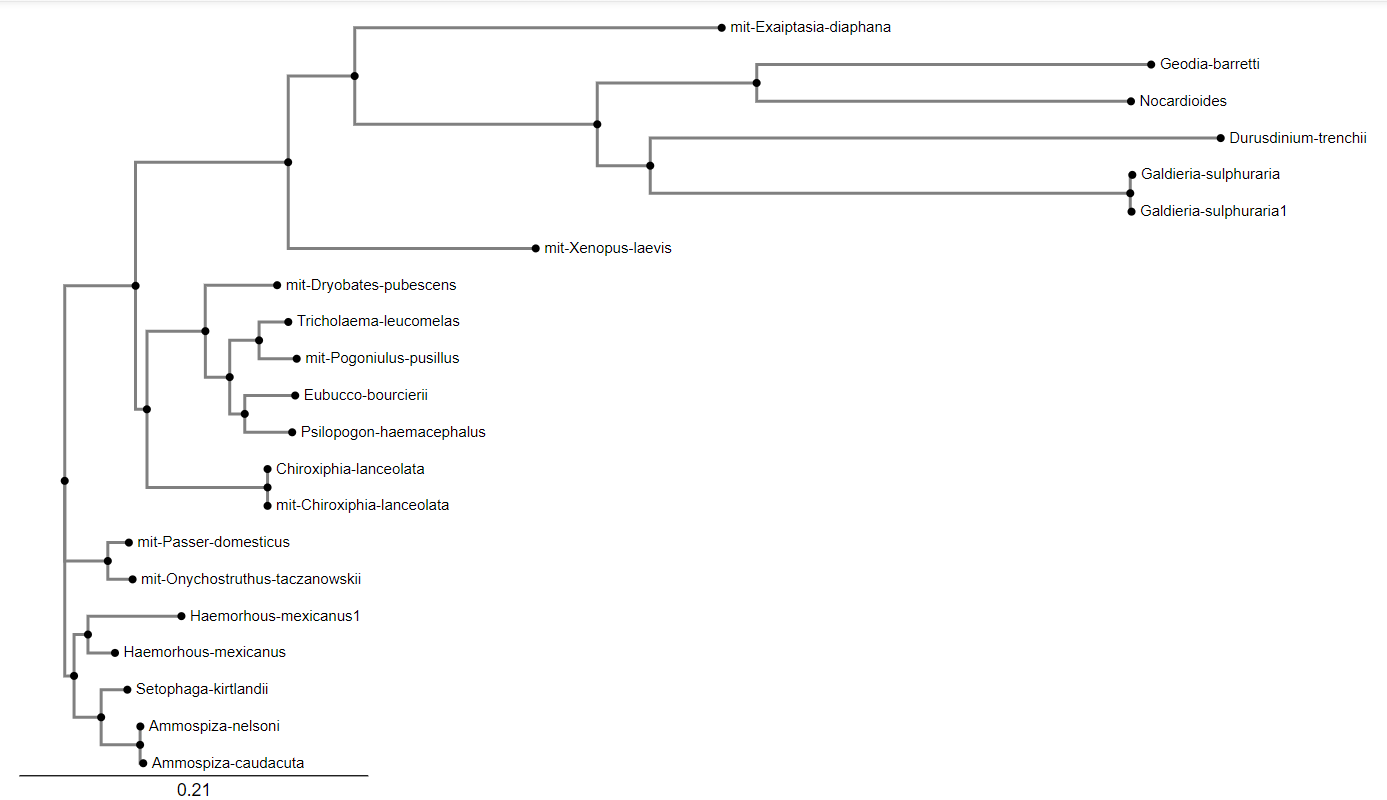 je pokazala, da je preiskovani protein soroden mitohondrijskim proteinom s podobno funkcijo (v imenu mit- na sliki). To kaže na sorodnost bakterij reda Nocardioides in bakterijskih prednikov mitohondrihev. Ta sorodnost je večja kot je sorodnost s proteini, ki imajo zapis v genomu evkariontov.
je pokazala, da je preiskovani protein soroden mitohondrijskim proteinom s podobno funkcijo (v imenu mit- na sliki). To kaže na sorodnost bakterij reda Nocardioides in bakterijskih prednikov mitohondrihev. Ta sorodnost je večja kot je sorodnost s proteini, ki imajo zapis v genomu evkariontov.
Najbolj in najmanj ohranjene regije#
Ohranjenost regij proteina je bila analizirana s poravnavo več zaporedij v orodju COBALT. Zaporedja, ki so bila poravnana, so najbolj podobna zaporedja iz nepresežnih baz podatkov. 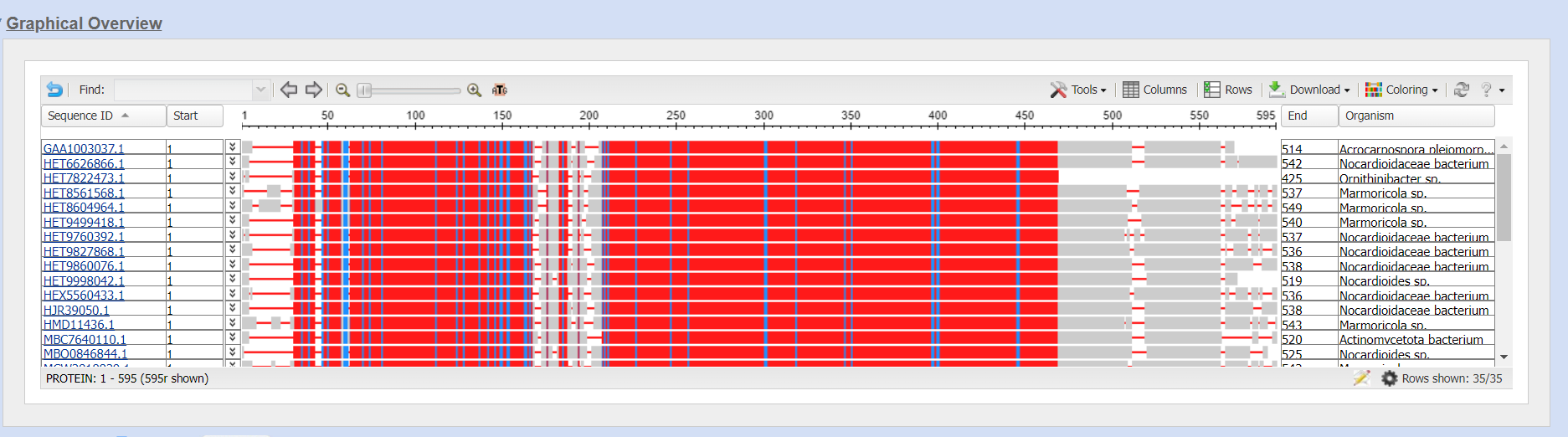 kažejo, da so najbolj ohranjene regije, regije, ki sovpadajo s proteinskimi domenami. Najbolj ohranjeni regiji se nahajta med aminoslinskimi ostanki 30 in 160 ter 210 in 460.
Prva regija sovpada s od AMP odvisno ligazno/sintetazno domeno, druga regija pa delno sovpada z AMP vezavno domeno. Zaporedje na delu 470-560 (brez 510-520) je ohranjeno, vendar manj kot prej omenjeni regiji. Ta del zaporedja sestavlja AMP vezavno domeno.
Tako kot pričakovano so najbolj ohranjeni deli proteina tisti, ki imajo vlogo pri sami funkciji proteina. Regije, ki niso ključne za funkcijo proteina so manj ohranjene - v primeru preiskovanega proteina so regije na N in C koncu proteina ter zaporedje med amiokislinskima ostankoma 510 in 520. Pri tej regiji gre najverjetneje za zanko, ki nima funkcjske vloge.
kažejo, da so najbolj ohranjene regije, regije, ki sovpadajo s proteinskimi domenami. Najbolj ohranjeni regiji se nahajta med aminoslinskimi ostanki 30 in 160 ter 210 in 460.
Prva regija sovpada s od AMP odvisno ligazno/sintetazno domeno, druga regija pa delno sovpada z AMP vezavno domeno. Zaporedje na delu 470-560 (brez 510-520) je ohranjeno, vendar manj kot prej omenjeni regiji. Ta del zaporedja sestavlja AMP vezavno domeno.
Tako kot pričakovano so najbolj ohranjeni deli proteina tisti, ki imajo vlogo pri sami funkciji proteina. Regije, ki niso ključne za funkcijo proteina so manj ohranjene - v primeru preiskovanega proteina so regije na N in C koncu proteina ter zaporedje med amiokislinskima ostankoma 510 in 520. Pri tej regiji gre najverjetneje za zanko, ki nima funkcjske vloge.
Za določanje najbolj in najmanj ohranjenih regij se lahko uporabi tudi orodje psi-blast. Ob uporabi tega orodja za preiskovan protein je bila vedno javljena  zaradi presežene uporabe procesorja na strežniku. Napaka se je pojavljala tudi ob spremenjenih vseh parametrih, ki bi zmanjšali uporabo procesorja. Analiza s psi-blast torej ni bila mogoča, zato se je potrebno glede ohranjenosti regij zanašati na rezultate poravnave zaporedij.
zaradi presežene uporabe procesorja na strežniku. Napaka se je pojavljala tudi ob spremenjenih vseh parametrih, ki bi zmanjšali uporabo procesorja. Analiza s psi-blast torej ni bila mogoča, zato se je potrebno glede ohranjenosti regij zanašati na rezultate poravnave zaporedij.
Podobni evkariontski proteini#
Bezoat-CoA ligazi, ki smo jo analizirali, najbolj podoben evkariontski protein je bezoat-CoA ligaza iz globokomorske spužve Geodia barretti, identičnost je 52 % (Per. Ident.). To kaže drugi zadetek pri 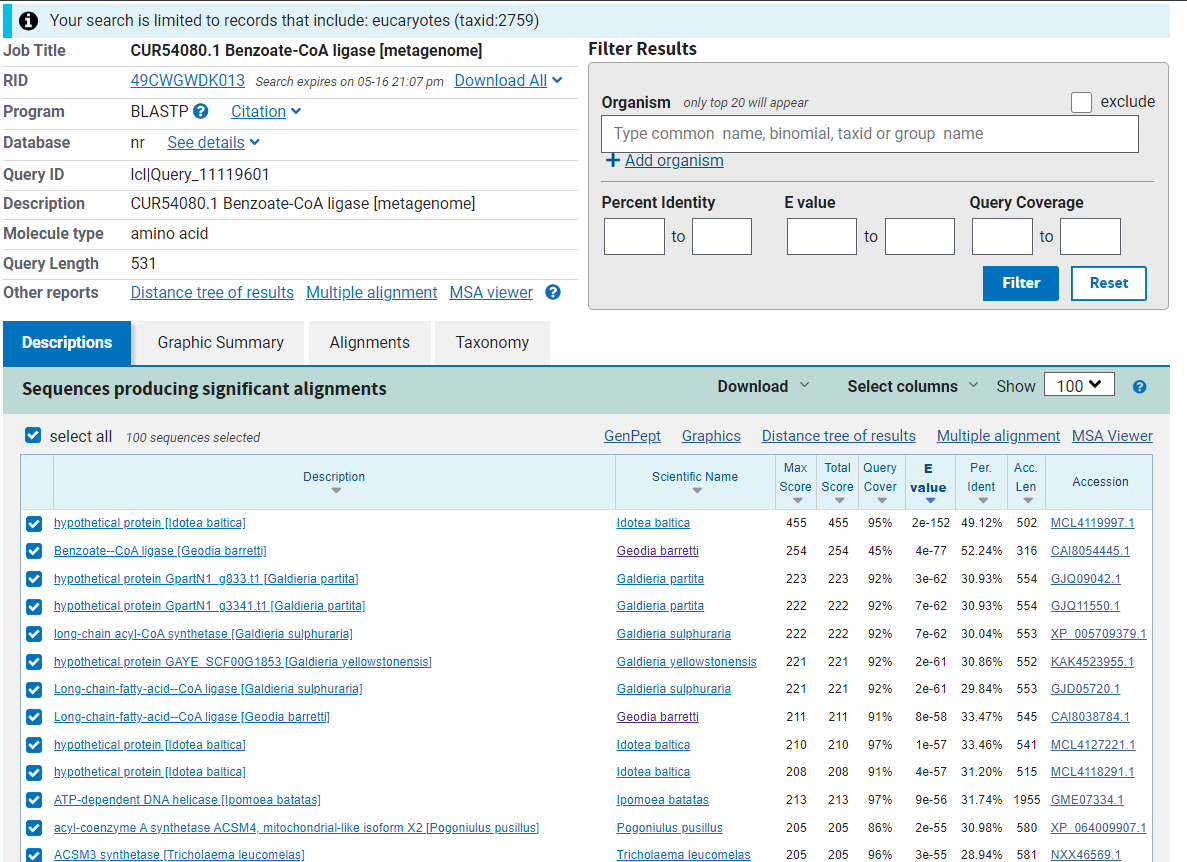 , kjer je iskanje zožano na ekvariontske organizme. Prvi zadetek iskanja v tem primeru ni protein, ki ga iščemo, saj je to hipotetični protein in njegov dejanski obstoj ni dokazan. Funkcija tega evkariontskega proteina iz spužve je enaka funkcji preiskovanega proteina, hkrati ima evkariontski protein prav tako od AMP odvisno ligazno domeno. To kaže na evolucijsko sorodnost obeh proteinov.
Ostali rezultati (brez hipotetičnih proteinov) iskanja kažejo, da imajo ti proteini podobno funkcijo kot preiskovani protein, in sicer ligacija druge molekule z CoA. Več manj podobnih zadetkov pri tem iskanju pa pripada zaporedjem za mitohondrijsko acil-CoA sintetazo pri različnih skupinah evariontov. Podobnost preiskovanega proteina je torej višja za prokatriontske proteine (na to kažejo tudi zadetki, da so mitohondrijski proteini bolj podobni kot ostali evkariontski) kot za evkariontske proteine, ki imajo zapis v genomu.
, kjer je iskanje zožano na ekvariontske organizme. Prvi zadetek iskanja v tem primeru ni protein, ki ga iščemo, saj je to hipotetični protein in njegov dejanski obstoj ni dokazan. Funkcija tega evkariontskega proteina iz spužve je enaka funkcji preiskovanega proteina, hkrati ima evkariontski protein prav tako od AMP odvisno ligazno domeno. To kaže na evolucijsko sorodnost obeh proteinov.
Ostali rezultati (brez hipotetičnih proteinov) iskanja kažejo, da imajo ti proteini podobno funkcijo kot preiskovani protein, in sicer ligacija druge molekule z CoA. Več manj podobnih zadetkov pri tem iskanju pa pripada zaporedjem za mitohondrijsko acil-CoA sintetazo pri različnih skupinah evariontov. Podobnost preiskovanega proteina je torej višja za prokatriontske proteine (na to kažejo tudi zadetki, da so mitohondrijski proteini bolj podobni kot ostali evkariontski) kot za evkariontske proteine, ki imajo zapis v genomu.
Model strukture#
Za benzoat-CoA ligazo iz bakterij rodu Nocardioides ni eksperimentalno določene strukture. Pri analizi strukture tega proteina si lahko pomagamo s strukturami podobnih proteinov.
Podobni proteini#
Iskanje podobnih proteinov z določeno strukturo je potekalo z orodjem blastp. Izbrana baza podatkov je bila pdb, zato da so bili kot zadetki prikazani samo proteini z že poznano strukturo.  so prav tako benzoat-CoA ligaze vendar iz drugih organizmov. Ti organizmi spadajo med prokarionte.
Če naredimo
so prav tako benzoat-CoA ligaze vendar iz drugih organizmov. Ti organizmi spadajo med prokarionte.
Če naredimo 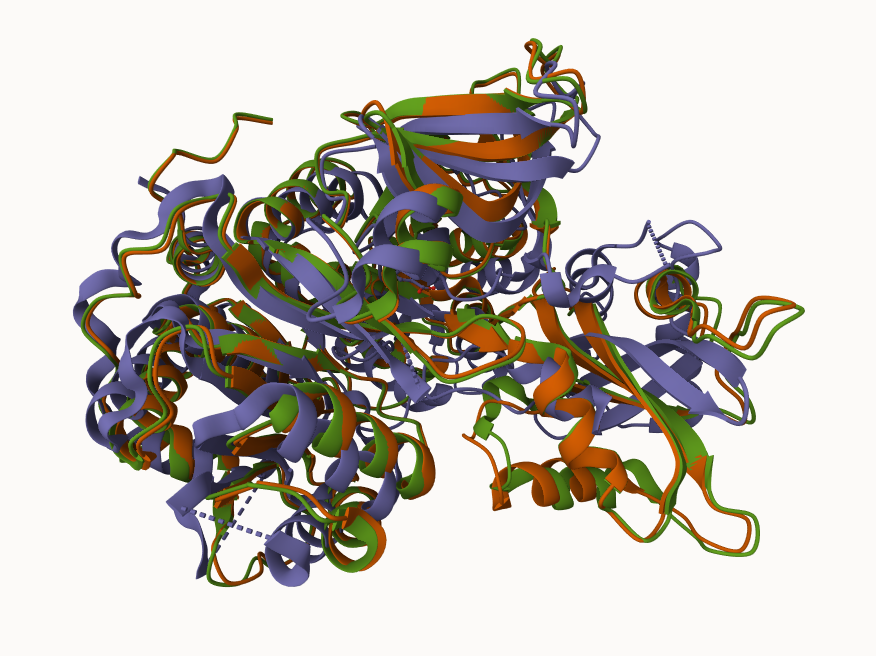 treh najbolj podobnih proteinov ugotovimo, da so strukture med seboj zelo podobne in da se vsi elementi sekundarne strukture povpadajo med seboj. Glede na zelo visoko podobnost struktur teh treh proteinov lahko sklepamo, da je najverjetneje taka tudi struktura preiskovanega proteina.
treh najbolj podobnih proteinov ugotovimo, da so strukture med seboj zelo podobne in da se vsi elementi sekundarne strukture povpadajo med seboj. Glede na zelo visoko podobnost struktur teh treh proteinov lahko sklepamo, da je najverjetneje taka tudi struktura preiskovanega proteina.
Če iskanje zožamo samo na 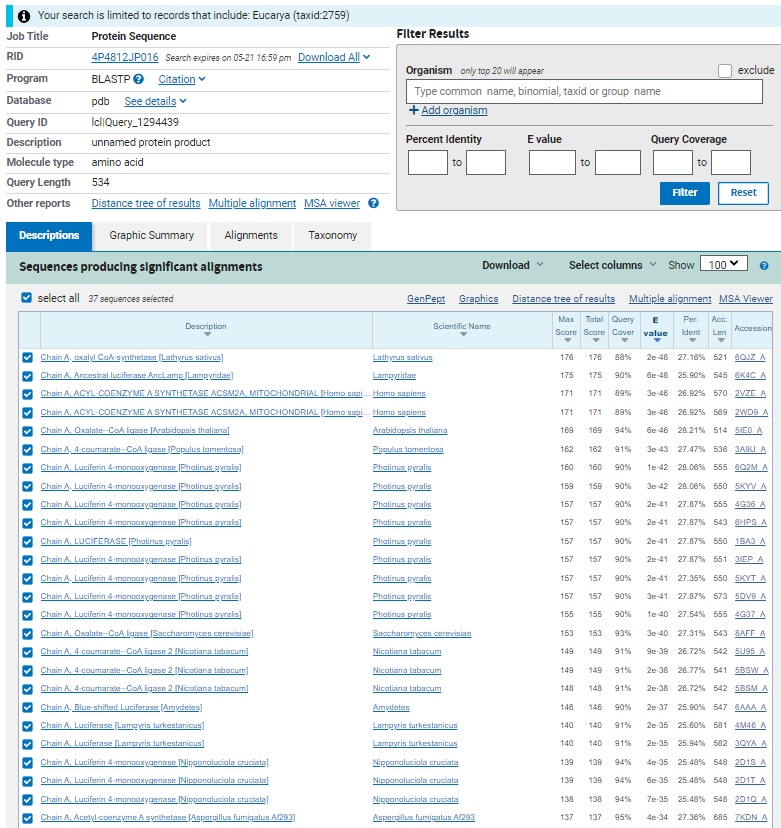 , je prvi zadetek (najbolj podoben) veriga A oksalil-CoA sintetaze iz indijskega graha (Lathyrus sativus). Glede na podatke na UniProt je
, je prvi zadetek (najbolj podoben) veriga A oksalil-CoA sintetaze iz indijskega graha (Lathyrus sativus). Glede na podatke na UniProt je 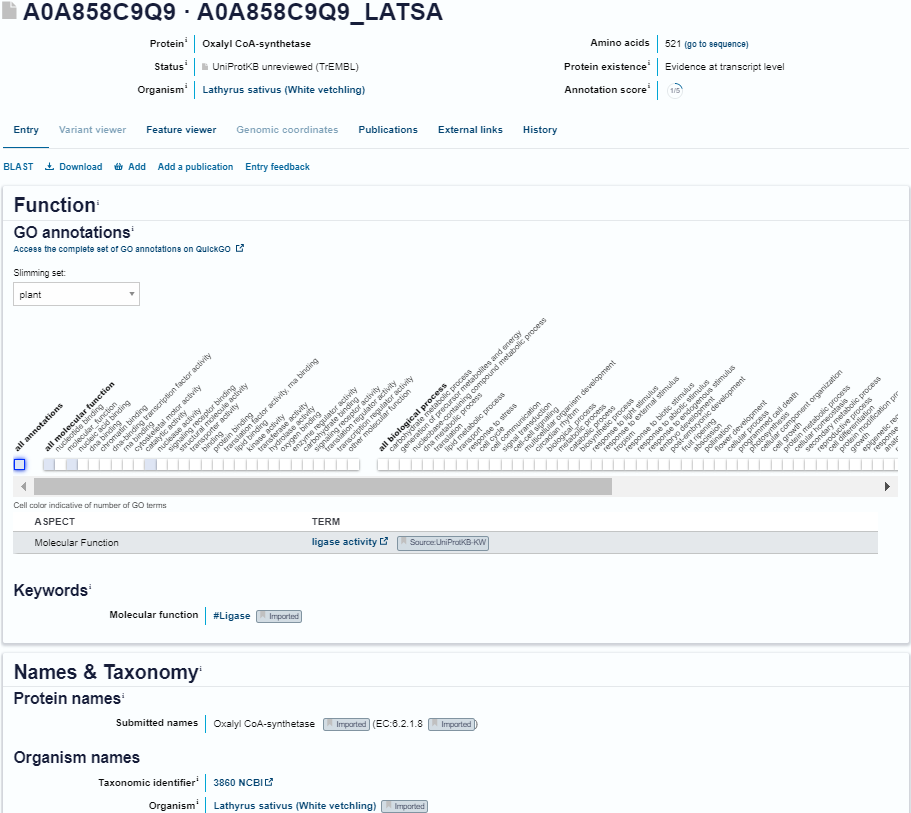 tega proteina ligacija oksalata s CoA ob porabi ATP. Funkcija je podobna funkciji preiskovanega proteina. To kaže na možnost evolucijske sorodnosti med tem proteinom in preiskovanim proteinom.
tega proteina ligacija oksalata s CoA ob porabi ATP. Funkcija je podobna funkciji preiskovanega proteina. To kaže na možnost evolucijske sorodnosti med tem proteinom in preiskovanim proteinom.
Če primerjamo strukturi preiskovanemi proteinu najbolj podobnega prokariontskega in evkatriontskega proteina z znano strukturo (rezultati iskanj opisani zgoraj), ugotovimo, da sta si strukturi precej podobni. Analiza je bila izvedena v programu Mol* s 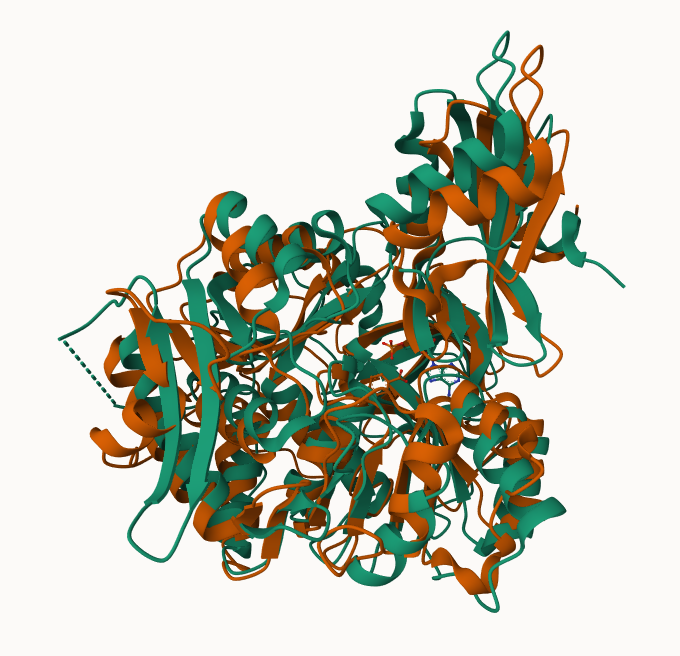 izbranih struktur - kot prokariontski protein (oranžna struktura na sliki) je benzoat-CoA ligaza iz organizma Rhodopseudomonas palustris, kot evkariontski protein pa oksalil-CoA sintetaza iz Lathyrus sativus.
Sekundarne strukture obeh proteinov se nahajo na enakih oziroma zelo podobnih pozicijah. Največje razlike med strukturama so v neurejenih delih proteina kot so zanke.
izbranih struktur - kot prokariontski protein (oranžna struktura na sliki) je benzoat-CoA ligaza iz organizma Rhodopseudomonas palustris, kot evkariontski protein pa oksalil-CoA sintetaza iz Lathyrus sativus.
Sekundarne strukture obeh proteinov se nahajo na enakih oziroma zelo podobnih pozicijah. Največje razlike med strukturama so v neurejenih delih proteina kot so zanke.
AlphaFold#
Z uprabo AlphaFold3 je bil pripravljen 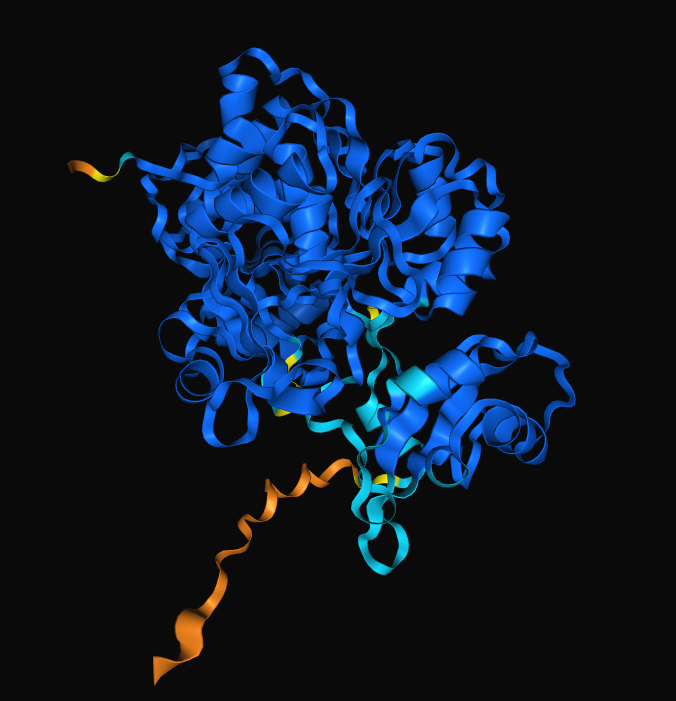 tega proteina. Modra obarvanost skoraj celotnega modela kaže na visoko verjetnost, da je taka tudi dejanska struktura tega proteina. Z primerjavo že določenih struktur podobnih proteinov s AlphaFold3 modelov ugotovimo, da strukture nimajo opaznih razlik. Tudi ta primerjava kaže na to, da AlphaFold3 model zelo verjetno dobro napove strukturo proteina, vendar bi bila za potrditev potrebna eksperimentalna določitev strukture.
Model strukture kaže, da je najmanj zanesljiv del modela N-konec proteina. To je pričakovano glede na to, da so N-konci ekperimentalno določenih struktur pogosto nedoločeni zaradi večje gibljivosti tega dela proteina.
tega proteina. Modra obarvanost skoraj celotnega modela kaže na visoko verjetnost, da je taka tudi dejanska struktura tega proteina. Z primerjavo že določenih struktur podobnih proteinov s AlphaFold3 modelov ugotovimo, da strukture nimajo opaznih razlik. Tudi ta primerjava kaže na to, da AlphaFold3 model zelo verjetno dobro napove strukturo proteina, vendar bi bila za potrditev potrebna eksperimentalna določitev strukture.
Model strukture kaže, da je najmanj zanesljiv del modela N-konec proteina. To je pričakovano glede na to, da so N-konci ekperimentalno določenih struktur pogosto nedoločeni zaradi večje gibljivosti tega dela proteina.
