VAST+ - iskanje podobnih makromolekulskih kompleksov#
Avtorja: Ana Maučec, Gašper Možina
Datum predstavitve: 2022-05-18
Namen vaje#
Namen vaje je poiskati makromolekule s podobnimi 3D strukturami s programom VAST+ s poudarkom na podobni biološki enoti in rezultate primerjati z iskanjem zgolj z aminokislinskim zaporedjem z uporabo BLAST-a po zbirki pdb.
Program#
Program: VAST+
Avtorji programa: Madej T, Lanczycki CJ, Zhang D, Thiessen PA, Geer RC, Marchler-Bauer A, Bryant SH, National Center for Biotechnology Information, U.S. National Library of Medicine, National Institutes of Health (https://www.ncbi.nlm.nih.gov/)
Reference:
Madej, T., Lanczycki, C. J., Zhang, D., Thiessen, P. A., Geer, R. C., Marchler-Bauer, A., & Bryant, S. H. (2014) MMDB and VAST+: tracking structural similarities between macromolecular complexes. Nucleic acids research 42, D297–D303. [10.1093/nar/gkt1208] (https://doi.org/10.1093/nar/gkt1208)
Thomas Madej, Aron Marchler-Bauer, Christopher Lanczycki, Dachuan Zhang, Stephen H Bryant Biological Assembly Comparison With VAST Methods Mol. Biol. 2020(2112):175-186. [ 10.1007/978-1-0716-0270-6_13 ] (https://link.springer.com/protocol/10.1007/978-1-0716-0270-6_13)
Opis programa#
VAST+ (Vector Alignment Search Tool Plus) je programsko orodje namenjeno iskanju podobnih makromolekulskih kompleksov. Podobnosti med strukturami se izračunajo izključno na podlagi geometrijskih meril brez upoštevanja aminokislinskih zaporedij, kar orodju VAST+ omogoča prepoznavanje oddaljenih homologov. Zgrajen je na podlagi orodja VAST (Vector Alignment Search Tool), ki je namenjen iskanju proteinov in posameznih domen s podobno 3D strukturo. VAST+ njegove zmožnosti razširja, saj je z njim možno določiti strukture, ki imajo podobno oblikovane biološke enote. VAST+ torej uporabljamo za določevanje proteinskih kompleksov z enako ali podobno oligomerno strukturo, za primerjanje komformacij istega oligomera v različnem stanju (npr. oxy- in deoxyhemoglobin) in za primerjavo vezavnih vmesnih površin.
Algoritem VAST+ gradi na razultatih algoritma VAST, lahko pa bi temeljil tudi na kakšnem drugem programu za primerjavo struktur proteinov. VAST najprej identificira posamezne elemente sekundarne strukture in jih predstavi kot vektorje ter primerja matrike koordinat vektorjev med vsemi strukturami v MMDB (Molecular Modeling Database). VAST+ nato identificira vse možne pare proteinov iz ene in druge strukture in ju primerja. Nato izračuna kakšni koti rotacije so potrebni za superpozicijo teh dveh struktur in združi v skupke pare proteinov s podobnim kotom rotacije. Poravnavo največjega skupka uporabi za to, da generira superpozicijo oligomerov. Zatem izračuna vrednost RMSD (koren povprečne vrednosti kvadratov razlik). Izračunan RMSD uporabi kot prag za identifikacijo aminokislinskih ostankov z najbolj podobno 3D pozicijo, jih poravna in na podlagi tega kriterija prikaže superpozicijo primerjanih makromolekulskih kompleksov.
To orodje nam ne da zadetkov, če v strukturi ni prisotne nobene proteinske molekule ali ima ta manj kot tri sekundarne strukture, če struktura nima definirane biološke enote ali pa v primeru, da za kakšne na novo dodane strukture še niso določena VAST+ “sosedja”.
Vhodni podatki#
Kot vhodne podatke v program vnesemo idenetifikatorja Protein Data Bank (PDB) ID ali Molecular Modeling Databaste (MMDB) ID.


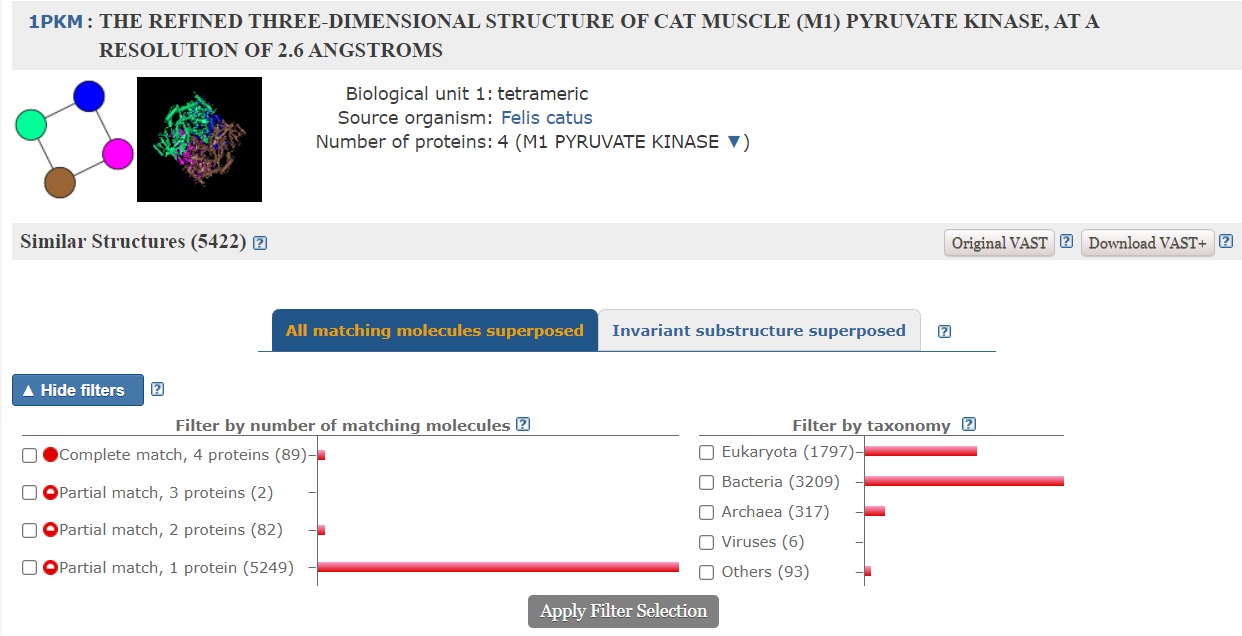
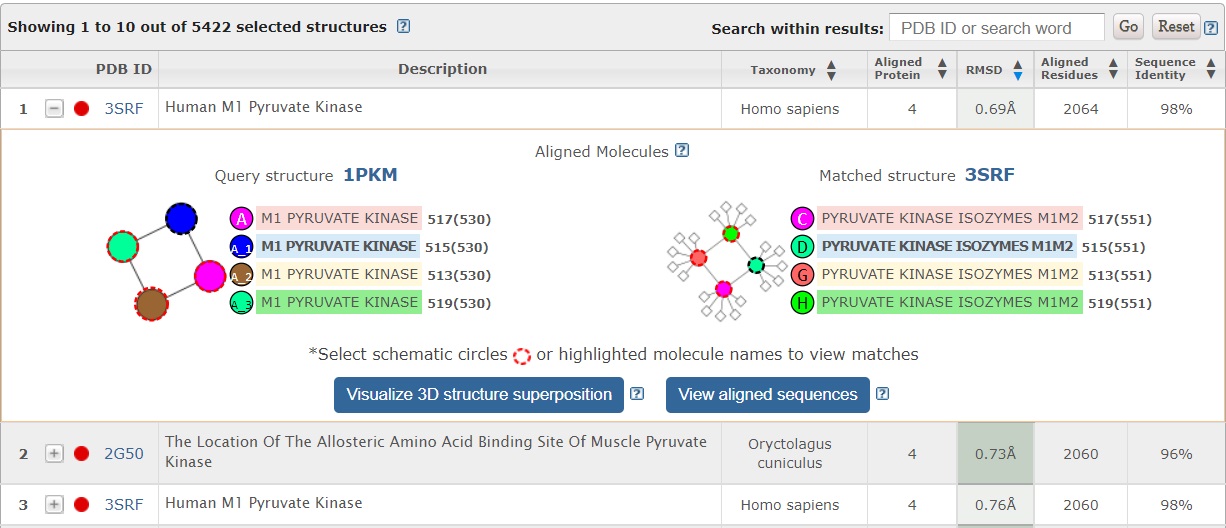
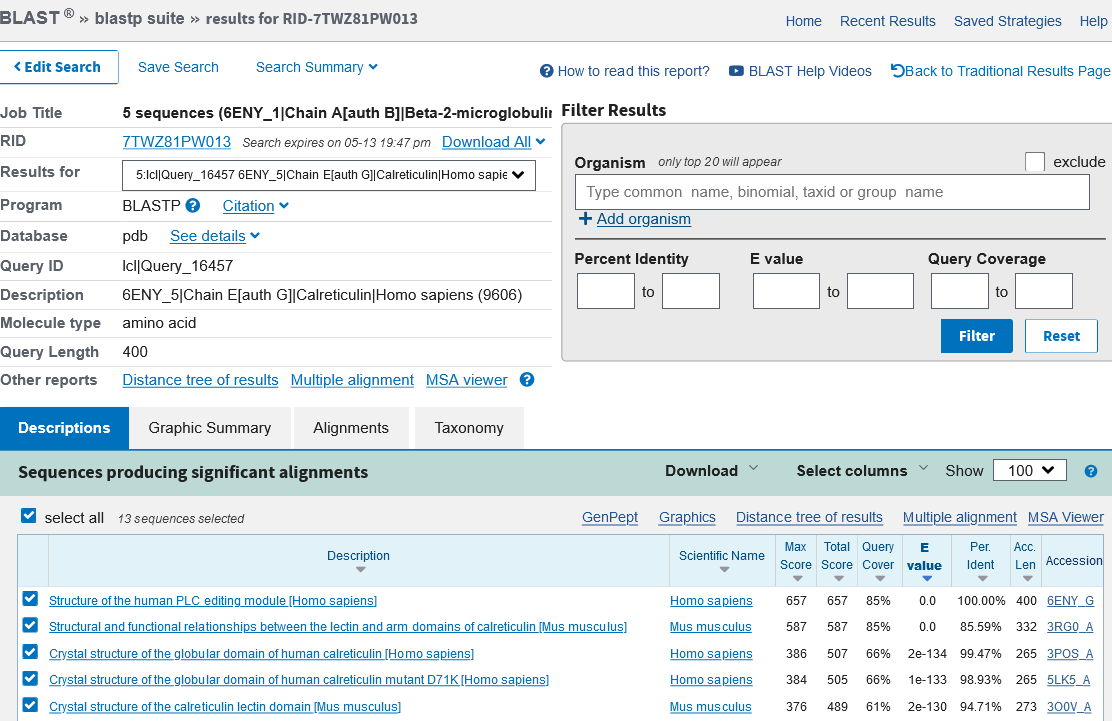
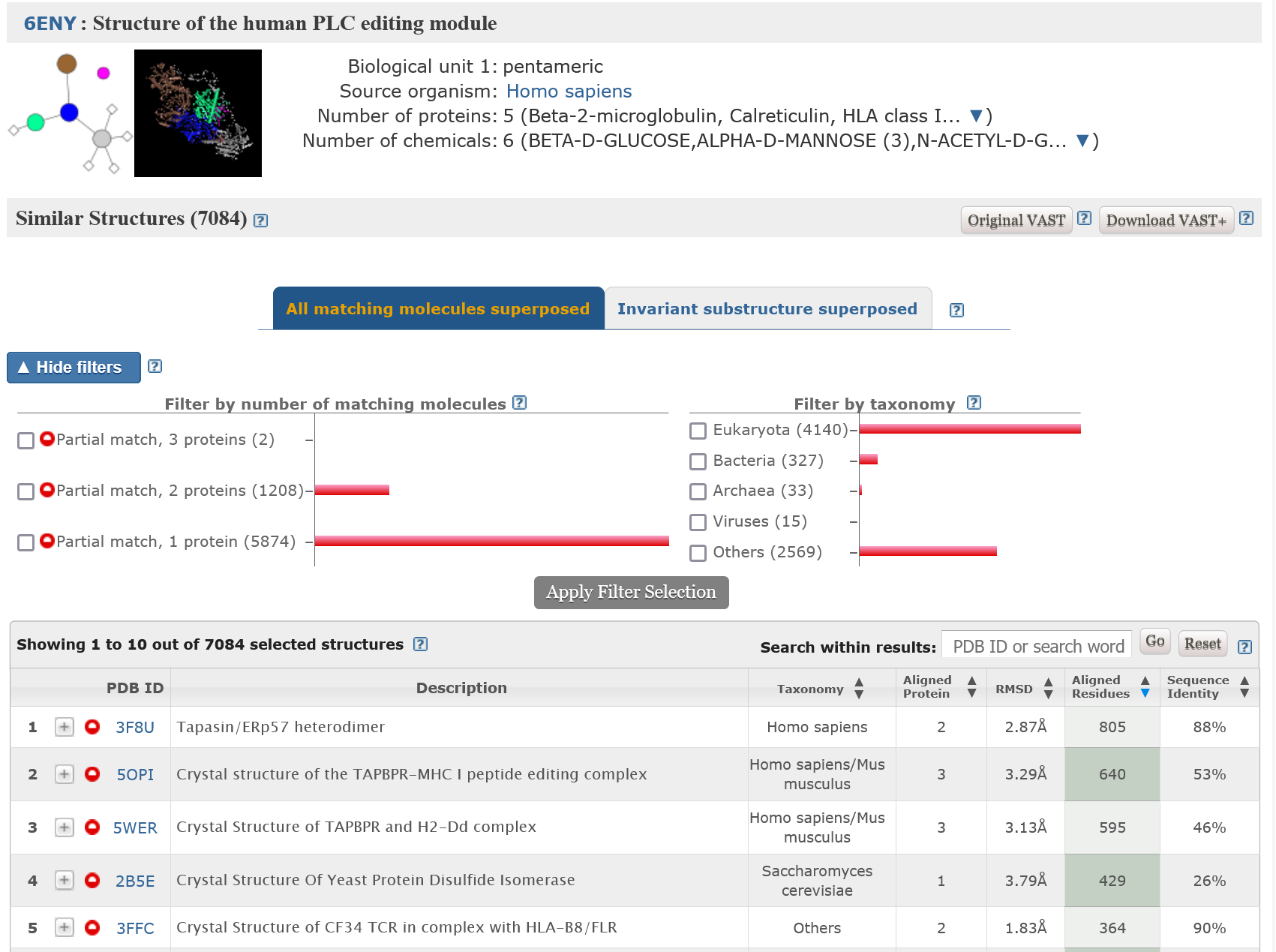
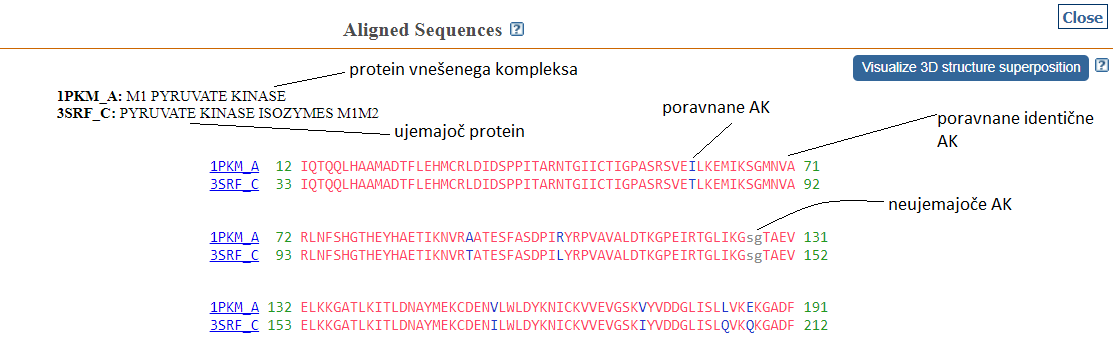 Vsak poravnan protein si lahko ogledamo tudi v 3D prostorski poravnavni (vse molekule/invariantne aminokisline), prikazani v programu iCn3D.
Vsak poravnan protein si lahko ogledamo tudi v 3D prostorski poravnavni (vse molekule/invariantne aminokisline), prikazani v programu iCn3D.