RNAfold - napoved sekundarnih RNA struktur#
Avtorja: Maj Priveršek, Metka Rus
Datum predstavitve: 2022-05-18
Namen vaje#
Ugotavljanje sekundarnih RNA struktur iz zaporedja nukleotidov s programom RNAfold
Program#
Program: RNAfold 2.5.0
Avtorji programa: Gruber AR, Lorenz R, Bernhart SH, Neuböck R, Hofacker IL.; Institute of Theoretical Chemistry, University of Vienna (http://www.tbi.univie.ac.at/)
Reference:
Lorenz, R. and Bernhart, S.H. and Höner zu Siederdissen, C. and Tafer, H. and Flamm, C. and Stadler, P.F. and Hofacker, I.L. “ViennaRNA Package 2.0”, Algorithms for Molecular Biology, 6:1 page(s): 26, 2011 The Vienna RNA Websuite. Nucleic Acids Research, Volume 36, Issue suppl_2, 1 July 2008, Pages W70-W74, DOI: 10.1093/nar/gkn188
Mathews DH, Disney MD, Childs JL, Schroeder SJ, Zuker M, Turner DH. (2004) Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure. Proc Natl Acad Sci U S A 101(19):7287-92, DOI: 101 (19) 7287-7292
Opis programa#
RNAfold je del obširnega serverja ViennaRNA Web Services, ki zajema mnoge različne načine za obdelavo in predvidevanje sekundarne strukture RNA in baze podatkov potrebne za njihovo delovanje. RNAfold Server je sposoben oblikovati sekundarno strukturo RNA iz zaporednja RNA nukleotidov. Deluje na podlagi matematičnega algoritma za napovedovanje sekundarnih struktur RNA, ki so ga predstavili Zuker et al. Gre za model na podlagi energije različnih vrst zank, ki se pojavljajo v sekundarnih strukturah.
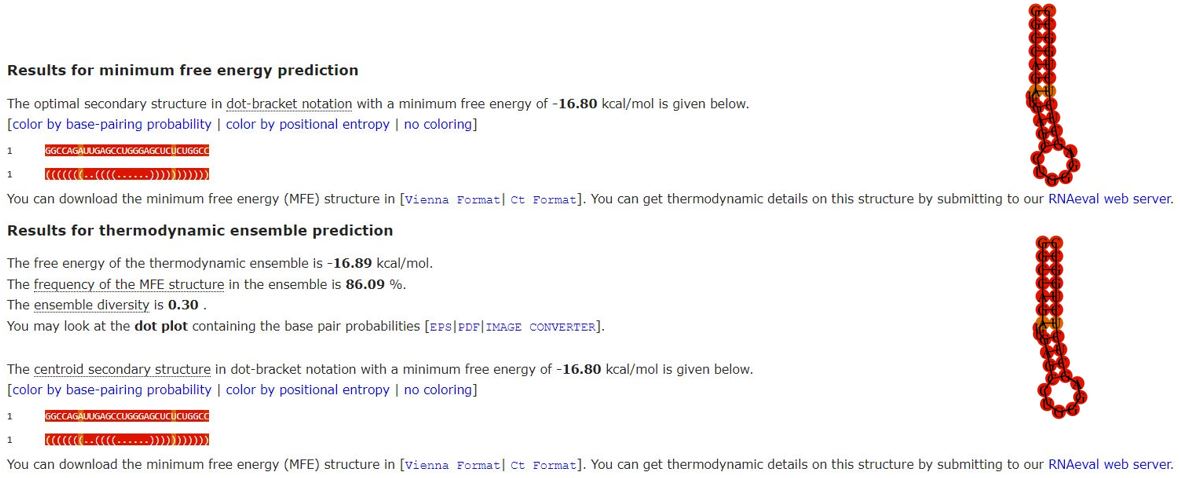
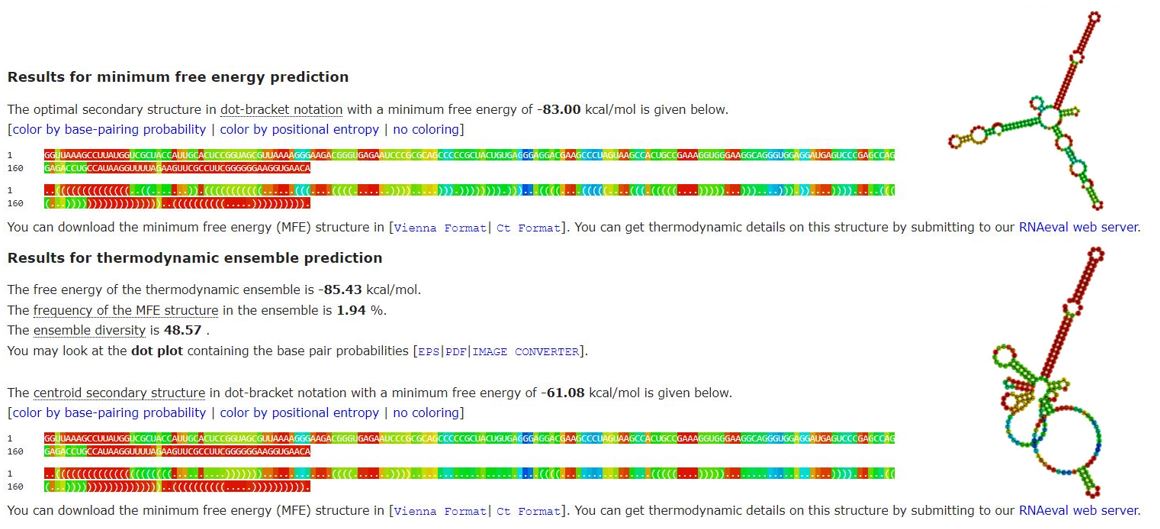
Algoritem za izračun oblike z najmanjšo energijo upošteva vse možne dovoljene oblike sekundarne strukture (vse oblike kjer je vsaka od baz vezana z maksimalno eno drugo bazo, vezi pa se med sabo ne križajo) in iz vseh teh izbere tisto z najmanjšo možno prosto energijo. To naredi na podlagi različnih parametrov: topološka in termodinamska pravila (določena v prej objavljenih člankih), dostopni eksperimentalni podatki o termoninamiki različnih oblik in motivov, znani podatki o odzivnosti nukleotidnih parov na kemijske modifikacije, o tem katere fospodiesterske vezi so najprej na udaru pri hidrolizi, podatki o ohranjenih sekundarnih strukturah in specifičnih dovoljenih daljnosežnih interakcijah med nukleotidi. Algoritem deluje na podlagi matematičnega modela, ki so ga predstavili že Nussinov et al., ki računa obliko z maksimalnim številom baznih parov. Algoritem je bil seveda prilagojen, da upošteva tudi vse prej naštete parametre.
Ker pa je RNA dinamična struktura, pa program podaja tudi energijo za ansamble vseh možnih dovoljenih struktur. Ansamble je set vseh sprejemljivih struktur RNA (pari se ne križajo in prekrivajo). Strukturni ansamble RNA ima Boltzmanovo porazdelitev, kjer je vrjetnost da se RNA nahaja v določeni obliki enaka enačbi \(P=\frac{e^{-β*εp}}{Z}\). V zapisu Z predstavlja particijsko funkcijo ali seštevek boltzmanovih funkcij za vse dovoljene oblike sekundarne strukture RNA. Cilj računanja je najti neko povprečno obliko v kateri se RNA nahaja, če vemo, da le-ta s časom spreminja svojo sekundarno obliko med vsemi možnimi dovoljenimi in je v določeni obliki delež časa proporcionalen s tem, kako energijsko ugodna je oblika. Program poda tudi strukturo, ki predstavlja srednjo/povprečno strukturo vseh struktur termodinamskega ansambla izračunanih s particijsko funkcijo.
Vhodni podatki#
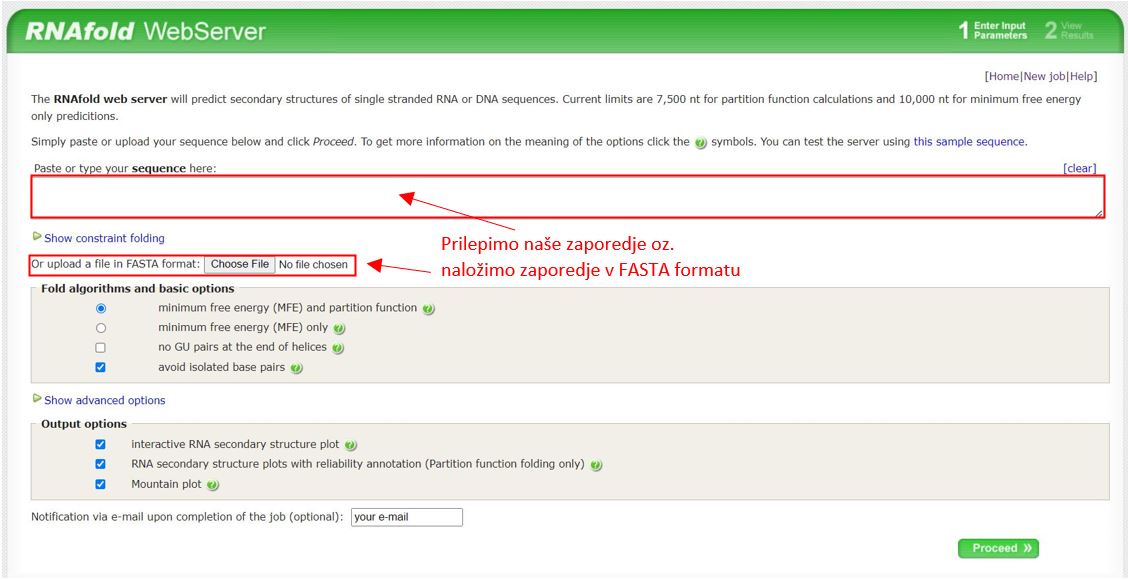
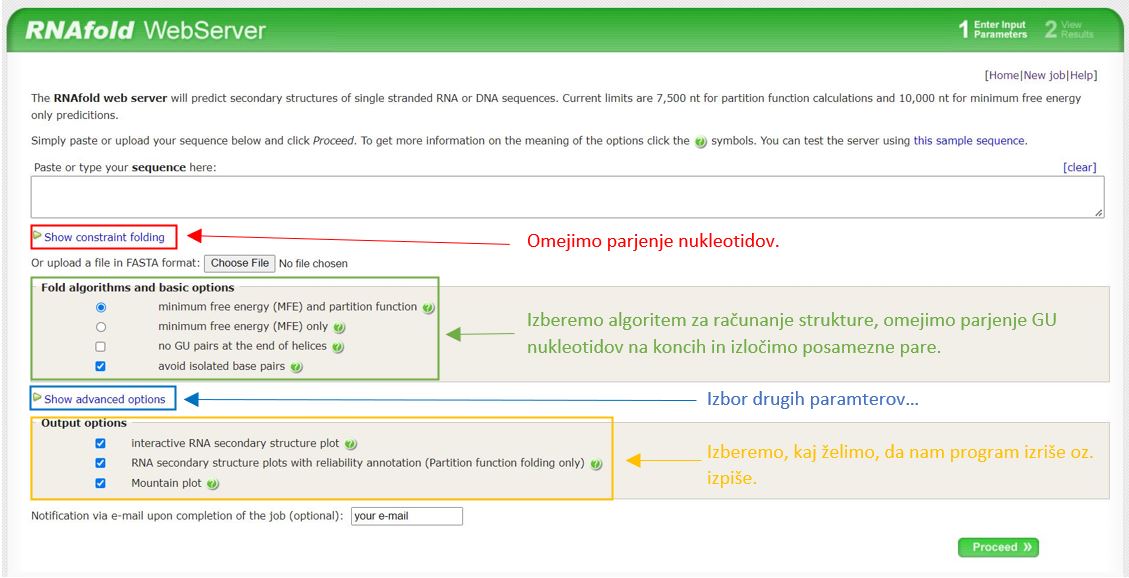
FASTA format RNA zaporedja ali samostojno RNA zaporedje