IUPred3#
Avtorja: Ana Marija Kodra, Neža Lanišek
Datum predstavitve: 2022-05-11
Namen vaje#
Spoznati spletno orodje IUPred3. S pomočjo tega orodja napovedati neurejene regije v proteinih in primerjati rezultate z eksperimentalnimi podatki.
Program#
Program: IUPred3
Avtorji programa: Gábor Erdős, Mátyás Pajkos, Zsuzsanna Dosztányi; Eötvös Loránd University
Reference:
Erdős, G.; Pajkos, M.; Dosztányi, Z. (2021) IUPred3: prediction of protein disorder enhanced with unambiguous experimental annotation and visualization of evolutionary conservation. Nucleic Acids Res. 49, 297–303. 10.1093/nar/gkab408
Dosztányi, Z. (2017) Prediction of protein disorder based on IUPred. Protein Sci. 27, 331–340. 10.1002/pro.3334
Dosztányi, Z.; Csizmók, V.; Tompa, P.; Simon, I. (2005) The pairwise energy content estimated from amino acid composition discriminates between folded and intrinsically unstructured proteins. J. Mol. Biol. 347, 827–839. 10.1016/j.jmb.2005.01.071
Opis programa#
Neurejene regije (IDPR; intrinsically disordered protein region) so regije v proteinih, ki nimajo ene same dobro definirane strukture, temveč lahko zavzamejo različna konformacijska stanja. Prisotne so v proteomu vseh organizmov, najbolj pa so razširjene pri evkariontih. Neurejene regije imajo pomembno funkcionalno vlogo pri mnogih celičnih procesih, predvsem pri signalizaciji in regulaciji celic, in so povezane z različnimi boleznimi. Sodelujejo tudi pri povezovanju različnih domen, sestavljanju makromolekularnih kompleksov in organizaciji nekaterih organelov. Nekatere neurejene regije proteinov lahko zavzamejo tudi urejeno strukturo – to lahko povzroči sprememba v redoks potencialu ali pa prisotnost urejene regije, s katero se neurejena regija poveže in tako tudi sama zavzame urejeno strukturo.
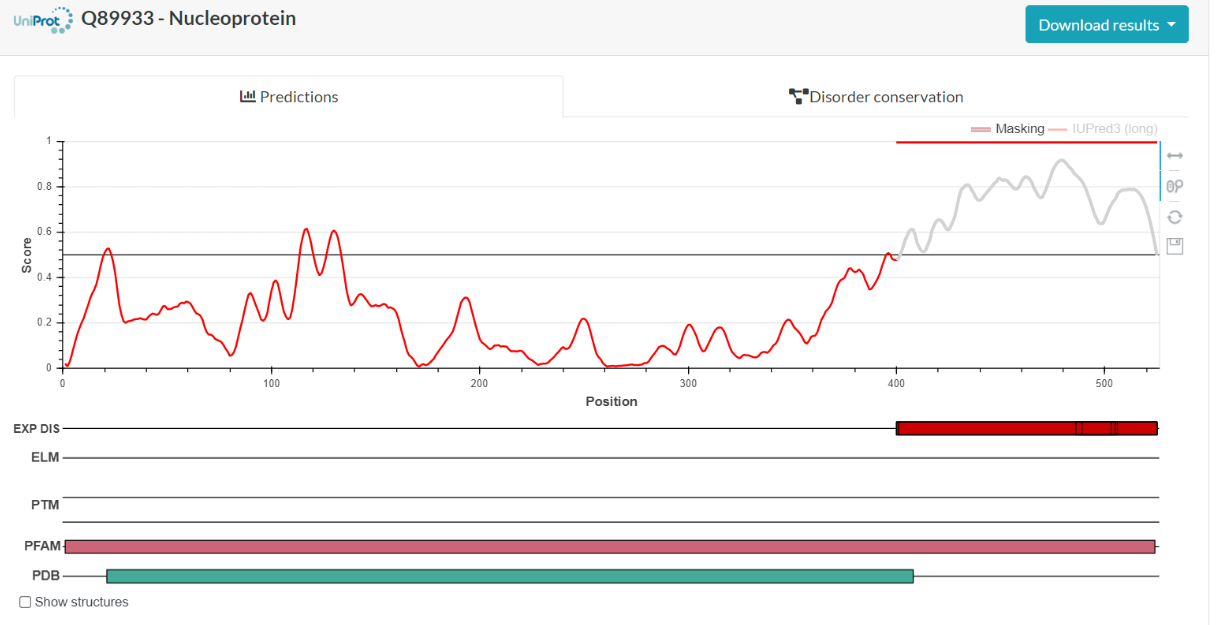
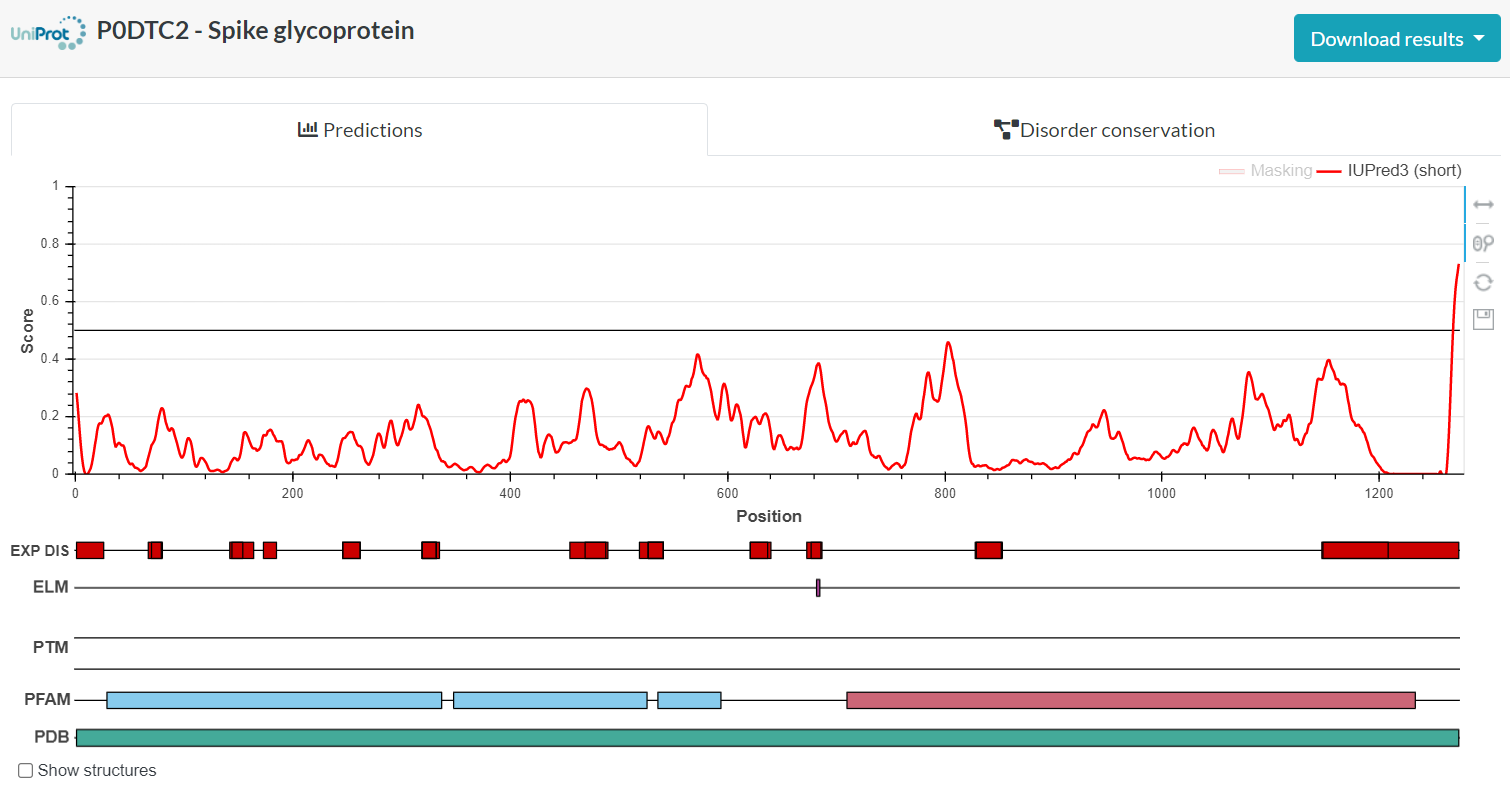
IUPred3 je program, ki združuje napoved neurejenih regij v proteinih z orodjem IUPred2 in napoved neurejenih vezavnih regij z orodjem ANCHOR2. Program pa lahko identificira tudi neurejene regije, ki pod določenimi pogoji lahko zavzamejo urejeno strukturo. Program je primeren za vse vhodne podatke, podane v pravi obliki, in ne glede na organizem, iz katerega prihaja protein, deluje enako.
IUPred3 temelji na metodi, ki oceni energijo interakcije med aminokislinskimi ostanki. Na podlagi znanih struktur globularnih proteinov se z uporabo interakcijske matrike, ki prikazuje parni statistični potencial, vsakemu aminokislinskemu ostanku dodeli vrednost energije na podlagi stikov, ki jih vzpostavi z drugimi aminokislinskimi ostanki v strukturi. Energije so ocenjene iz aminokislinskega zaporedja z uporabo matrike za oceno energije velikosti 20 × 20. Parametri v tej matriki so izračunani z metodo najmanjših kvadratov. Ocena energije interakcije je torej odvisna od vrste aminokislinskega ostanka in njegovih bližnjih sosedov. Osnovna predpostavka tega pristopa je, da so aminokislinski ostanki z ugodnimi energijami del urejene regije, medtem ko so aminokislinski ostanki z neugodnimi energijami del neurejene regije. Na koncu program ocenjeno vrednost energije interakcije pretvori v vrednost med 0 in 1. Za aminokislinske ostanke, ki jim program napove vrednost nad 0,5, je torej bolj verjetno, da so del neurejene regije, medtem ko je za aminokislinske ostanke, ki jim program napove vrednost pod 0,5, bolj verjetno, da so del urejene regije.
Vhodni podatki#
Kot vhodne podatke se v program lahko vnese accession code ali ID proteina iz UniProt-a, lahko pa se vnese aminokislinsko zaporedje v FASTA formatu ali naloži datoteka v FASTA formatu.