CATH: Protein Structure Classification Database#
Avtorja: Ivana Vukšinić, Zarja Weingerl
Datum predstavitve: 2022-05-11
Namen vaje#
Naučili se bomo uporabljati brskalnik CATH, ki na podlagi podatkov o aminokislinskem zaporedju in strukturi iskanega proteina, klasificira njegove domene in jih uvrsti v posamezne naddružine glede na njihovo evolucijsko sorodnost.
Program#
Program: CATH-Plus 4.3.0
Avtorji programa: I. Sillitoe, N. Dawson, T. Lewis, D. Lee, J. Lees, C. Orengo - Orengo Group, UCL
Reference:
Sillitoe, I.; Bordin, N.; Dawson, N.; Waman, V.P.; Ashford, P.; Scholes, H.M.; Pang, C.S.M.; Woodridge, L.; Rauer, C.; Sen, N.; Abbasian, M.; Le Cornu, S.; Lam, S.D.; Berka, K.; Varekova, I.H.; Svobodova, R.; Lees, J.; Orengo, C.A. (2021) CATH: increased structural coverage of functional space. Nucleic Acids Res. doi: 10.1093/nar/gkaa1079
Lewis, T.E.; Sillitoe, I.; Dawson, N.; Lam, S.D.; Clarke, T.; Orengo, C.A.; Lees, J.G. (2018) Gene3D: Extensive prediction of globular domains in proteins. Nucleic Acids Res. doi: 10.1093/nar/gkx1069
Opis programa#
Podatkovno zbirko CATH so ustvarili raziskovalci skupine Orengo na univerzi UCL, leta 1997. Ponuja informacije o evolucijskih odnosih med proteinskimi domenami.
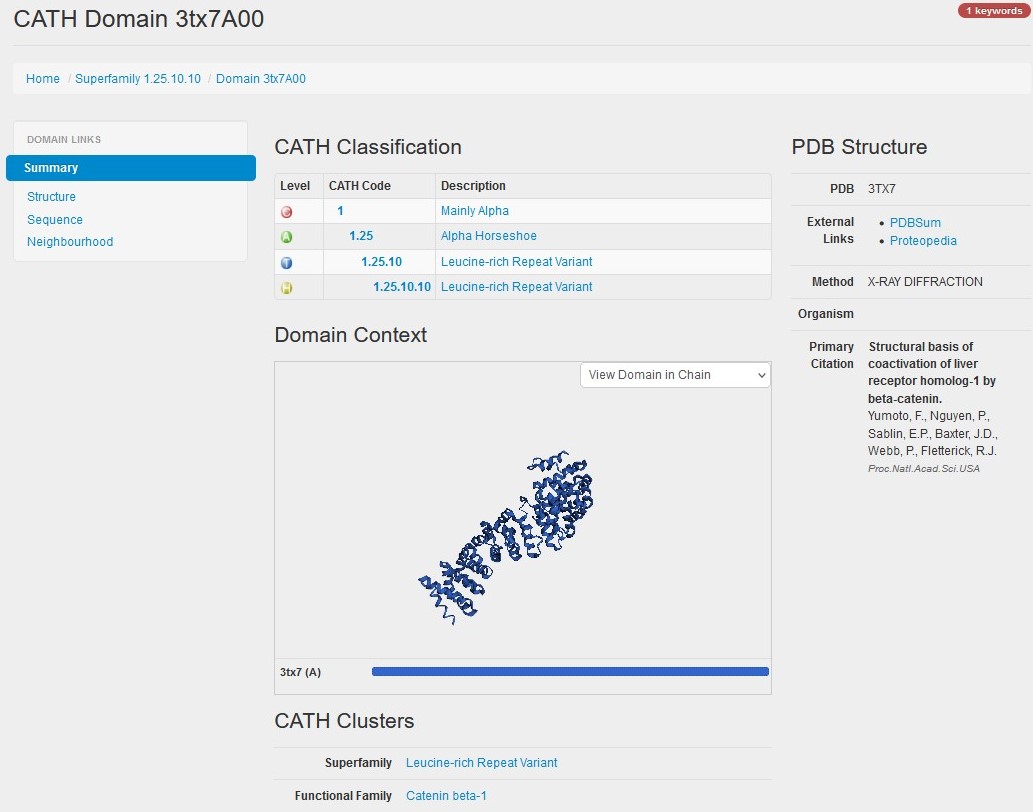
Uporablja semi-avtomatski postopek, s katerim 3D strukture, pridobljene iz podatkovne zbirke PDB, razcepi na polipeptidne verige, znotraj teh pa nato identificira posamezne domene. Te klasificira po naslednji hierarhiji:
C: Class (razvrščanje na podlagi sekundarne strukture: pretežno α, pretežno β, α/β, malo sekundarne strukture)
A: Architecture (razporeditev glede na orientacijo sekundarnih struktur v 3D prostoru)
T: Topology/fold (sosledje elementov sekundarnih struktur)
H: Homologous superfamily (razvrščanje glede na evolucijsko sorodnost).
Če najde iskani domeni evolucijsko sorodne (homologne) domene, jo uvrsti v ustrezno homologno naddružino. Poleg tega klasificira tudi domene znotraj proteinskih zaporedij z do sedaj nepotrjeno 3D strukturo. Podatke o zaporedjih pridobi s pomočjo pomožnega programa Gene3D.
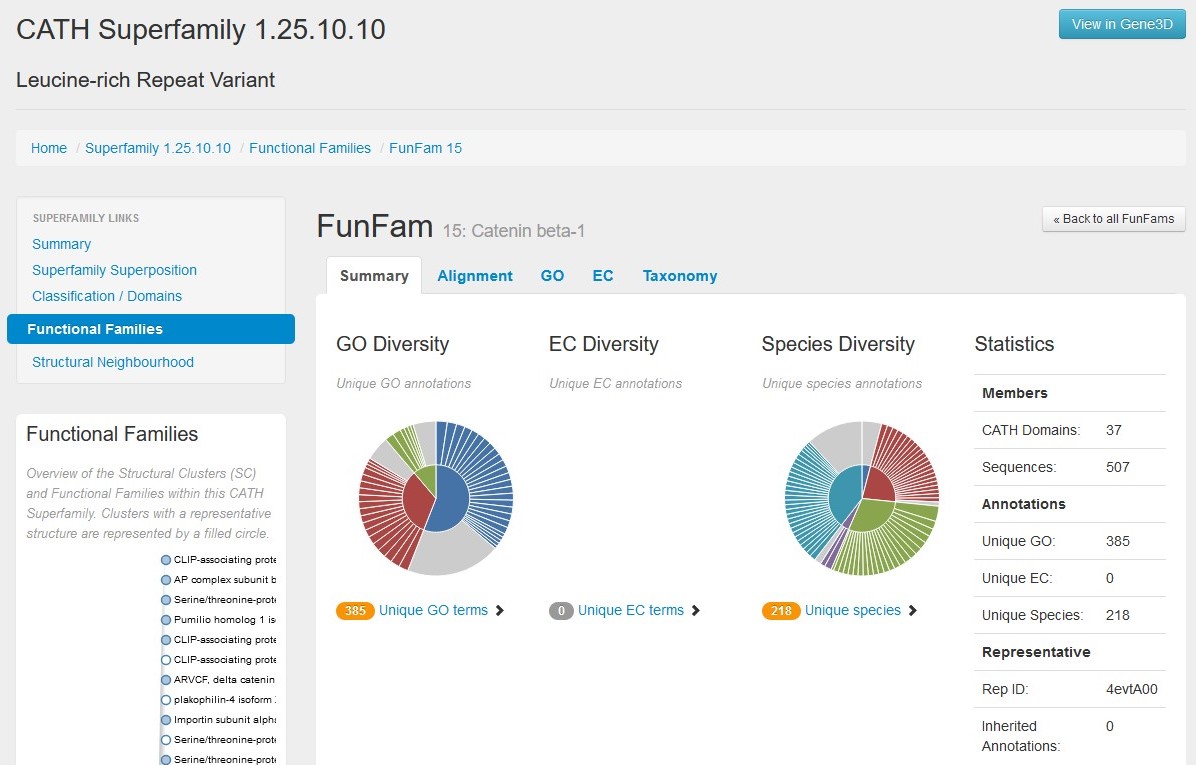
Z uporabo algoritma SSAP se za vsako naddružino izdela superpozicija vseh reprezentativnih proteinskih domen. Predstavniki iste naddružine si delijo ohranjeno strukturno jedro, kar pa ne pomeni, da opravljajo tudi enako funkcijo. Zato so razvili klasifikacijski protokol FunFams, ki dodatno razvršča naddružine v funkcionalno koherentne skupine. Predstavniki iste funkcionalne družine imajo podobno strukturo in funkcijo.
Vhodni podatki#
Ključne besede, PDB koda, GO termini, aminokislinsko zaporedje v FASTA formatu ali PDB datoteka.