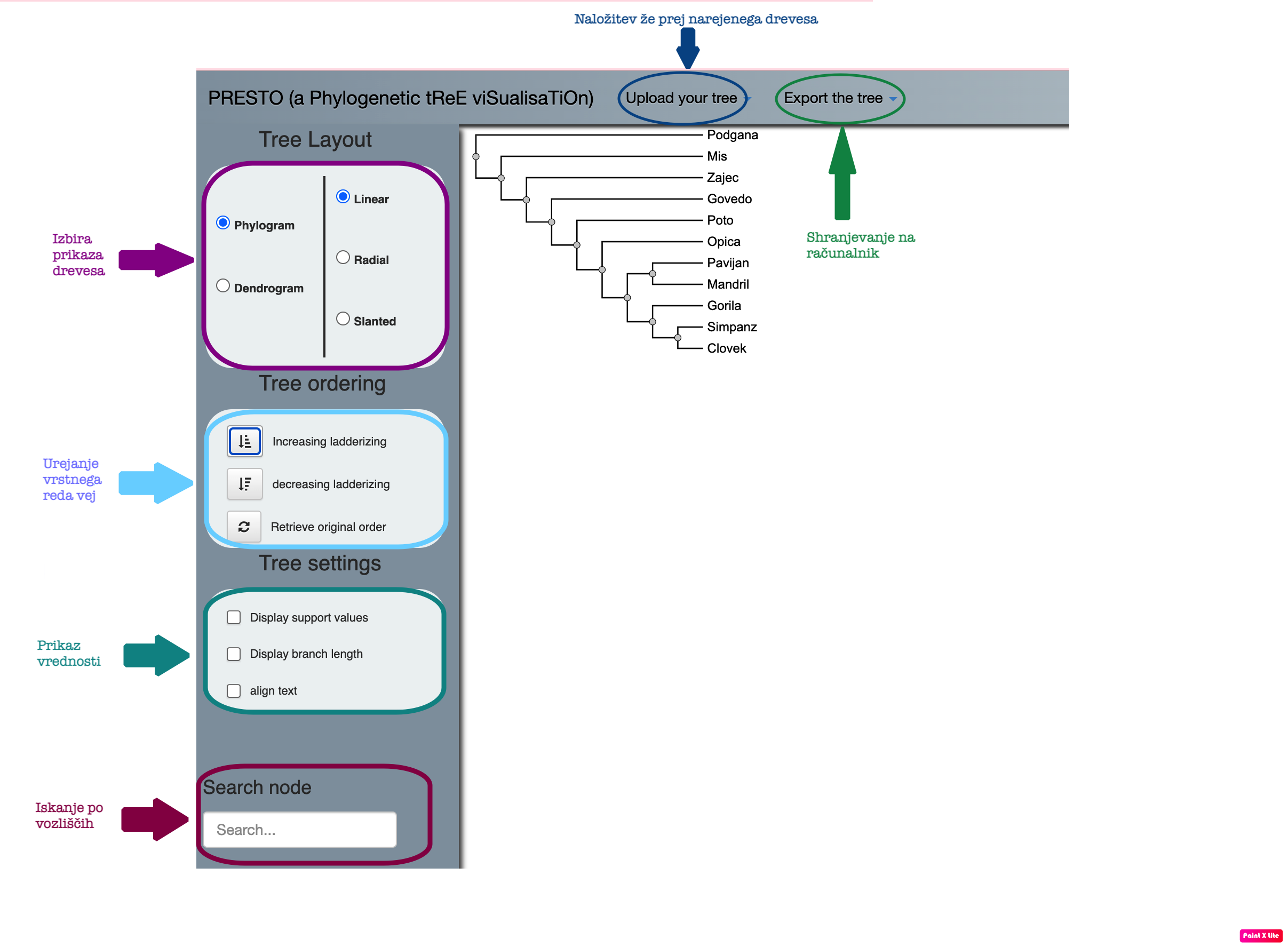
PhyML-filogenetska drevesa#
Avtorja: Maja Deutsch, Sara Jerič
Datum predstavitve: 2022-05-13
Namen vaje#
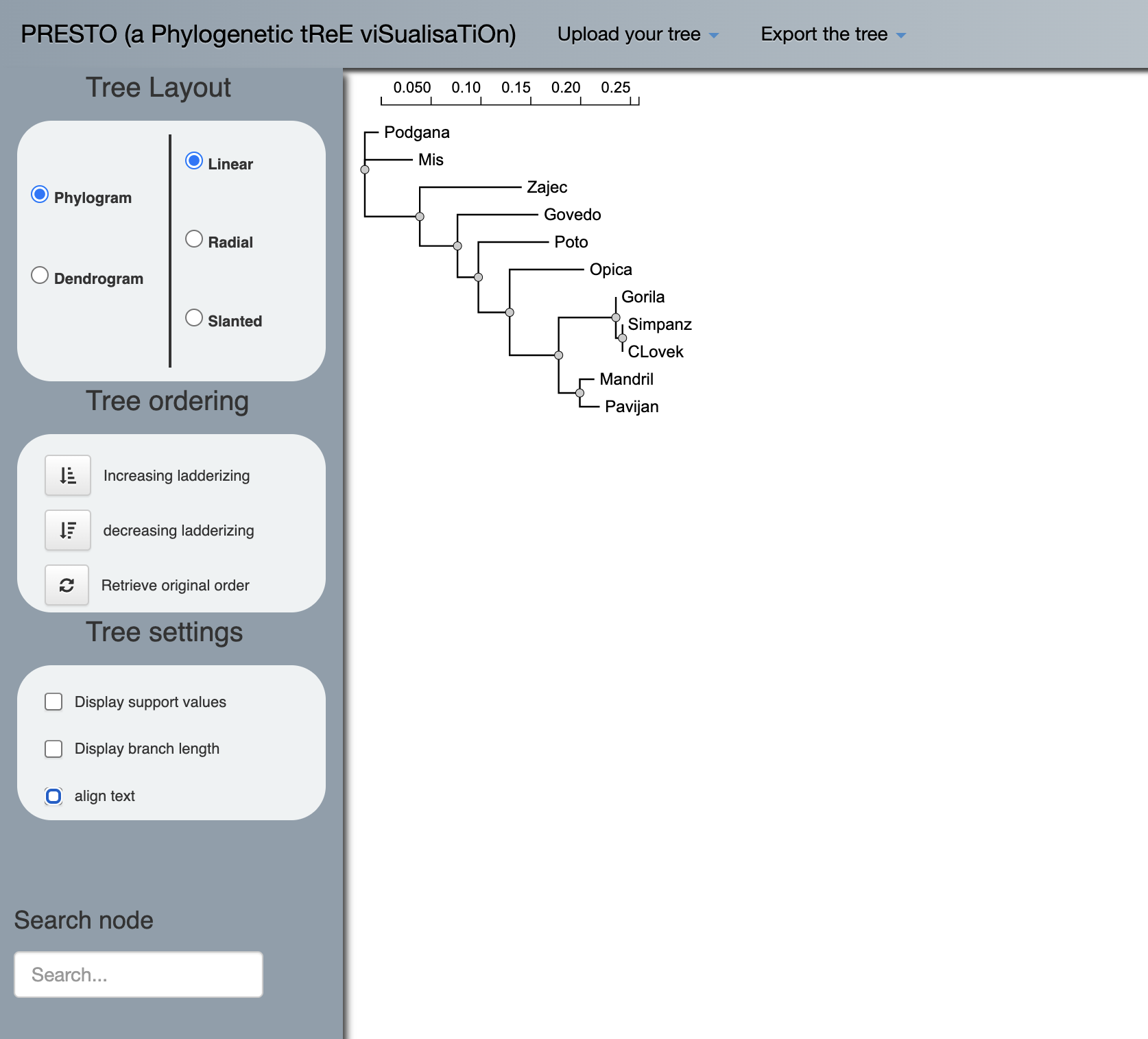
Na tej vaji bomo narisali filogenetsko drevo, ki bo temeljilo na principu največje verjetnosti.
Program#
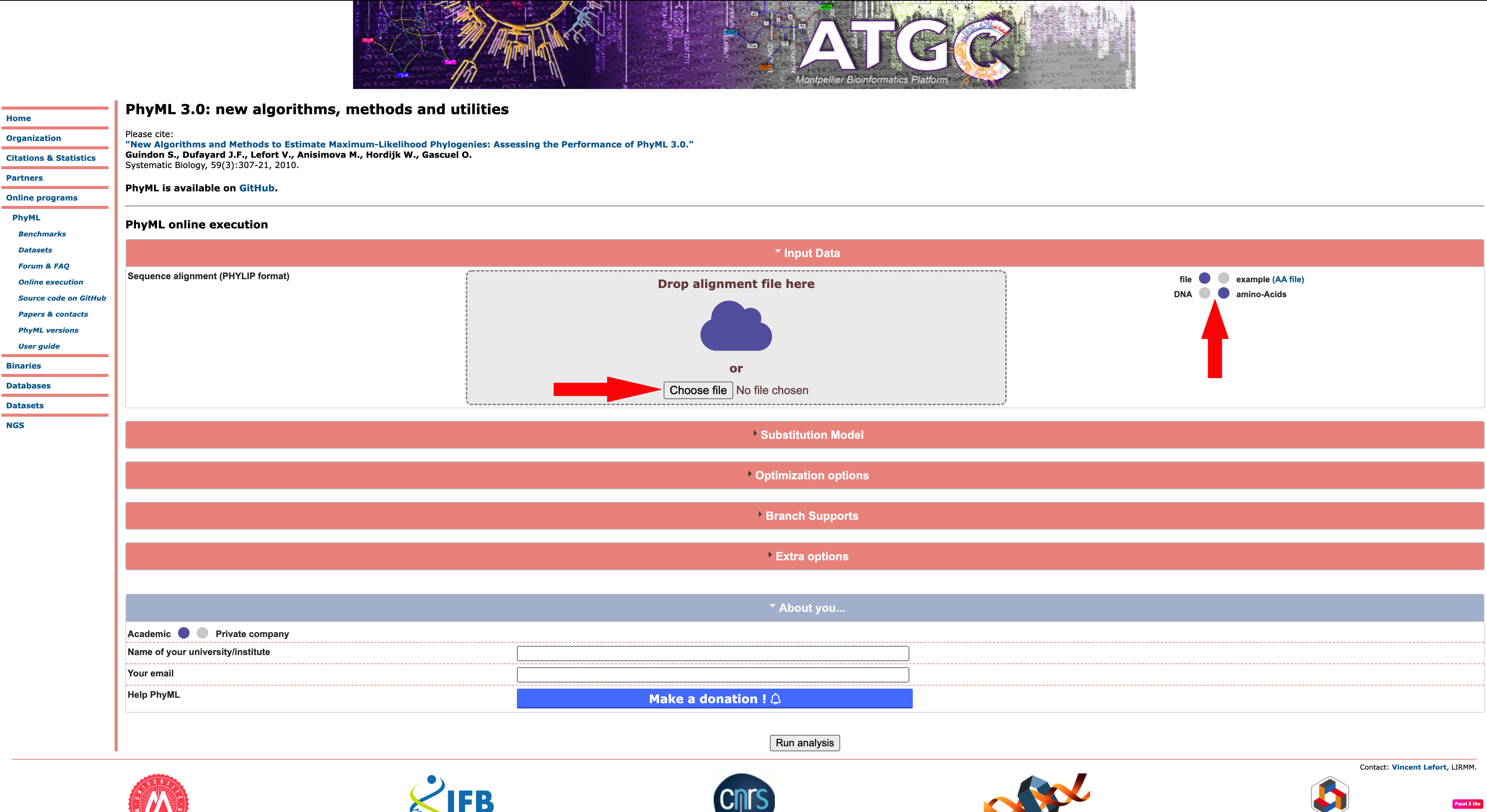
Program: PhyML 3.0
Avtorji programa: Guindon S., Dufayard J.F., Lefort V., Anisimova M., Hordijk W., Gascuel O., Institut Français de Bioinformatique in France Génomique
Reference:
Guindon, S.; Dufayard, J.; et al. (2010) New Algorithms and Methods to Estimate Maximum-Likelihood Phylogenies: Assessing the Performance of PhyML 3.0 Systematic Biology. 52(5),696-704. [10.1093/sysbio/syq010] (https://doi.org/10.1093/sysbio/syq010)
Manalastas-Cantos, K.; Konarev, P.V.; Hajizadeh, N.R.; Kikhney, A.G.; Petoukhov, M.V.; Molodenskiy, D.S.; Panjkovich, A.; Mertens, H.D.T.; Gruzinov, A.; Borges, C.; et al. (2021) ATSAS 3.0: Expanded Functionality and New Tools for Small-Angle Scattering Data Analysis. Journal of Applied Crystallograpy 54, 343–355. 10.1107/S1600576720013412
Opis programa#
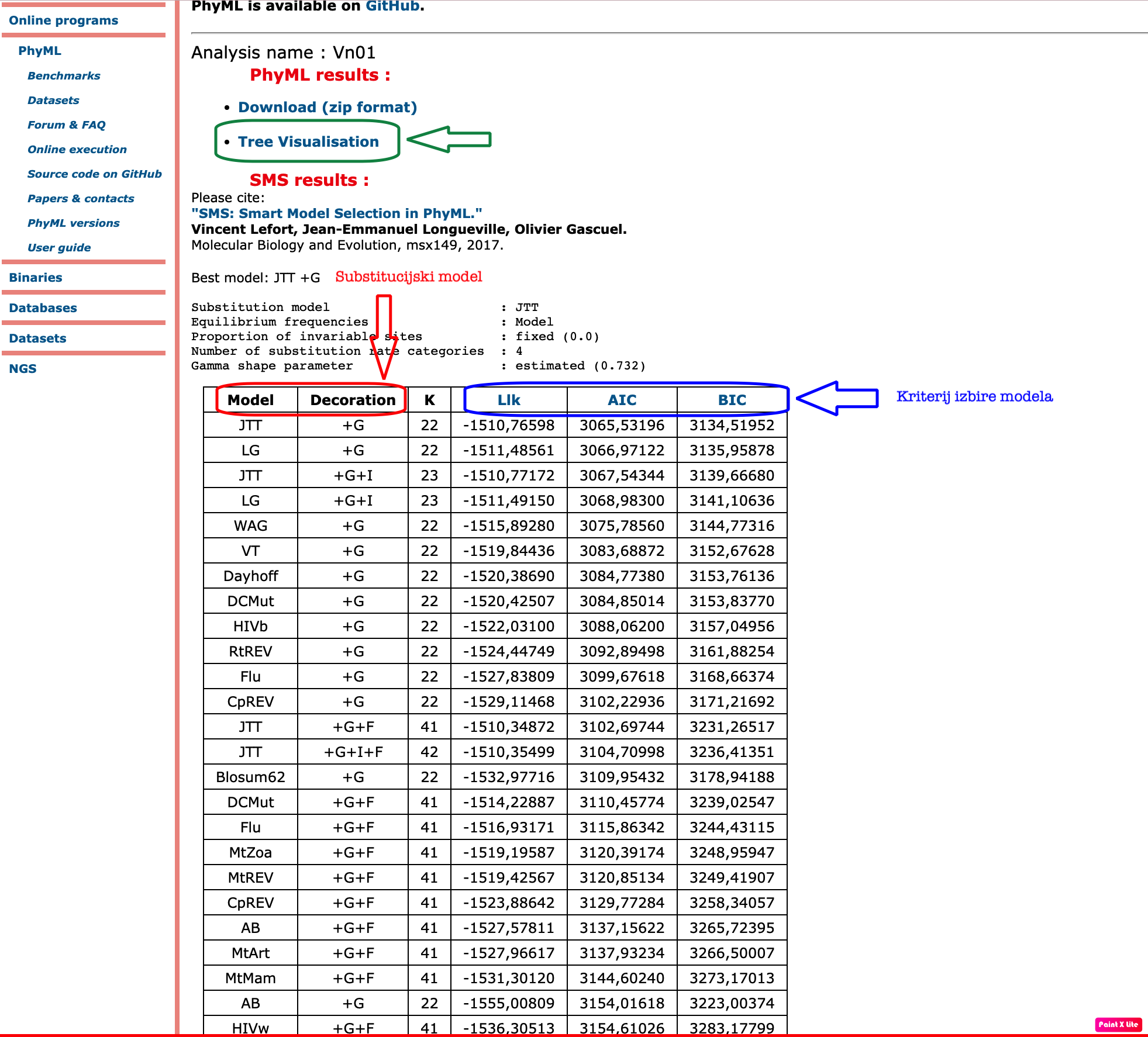
PhyML je program (software package), ki je primarno namenjen za oceno največje verjetnosti filogenije iz poravnav zaporedij. Ta zaporedja so lahko aminokislinska ali nukleotidna. Največja prednost PhyML je veliko število nadomestnih modelov, ki so povezani z različnimi možnostmi za iskanje topologij filogenetskega drevesa. PhyML deluje hitro do počasi, vendar je zelo natančen. PhyML s pomočjo testa razmerij na osnovi največje verjetnosti poišče najbolj podobne organizme in na podlagi tega izriše filogenetsko drevo.
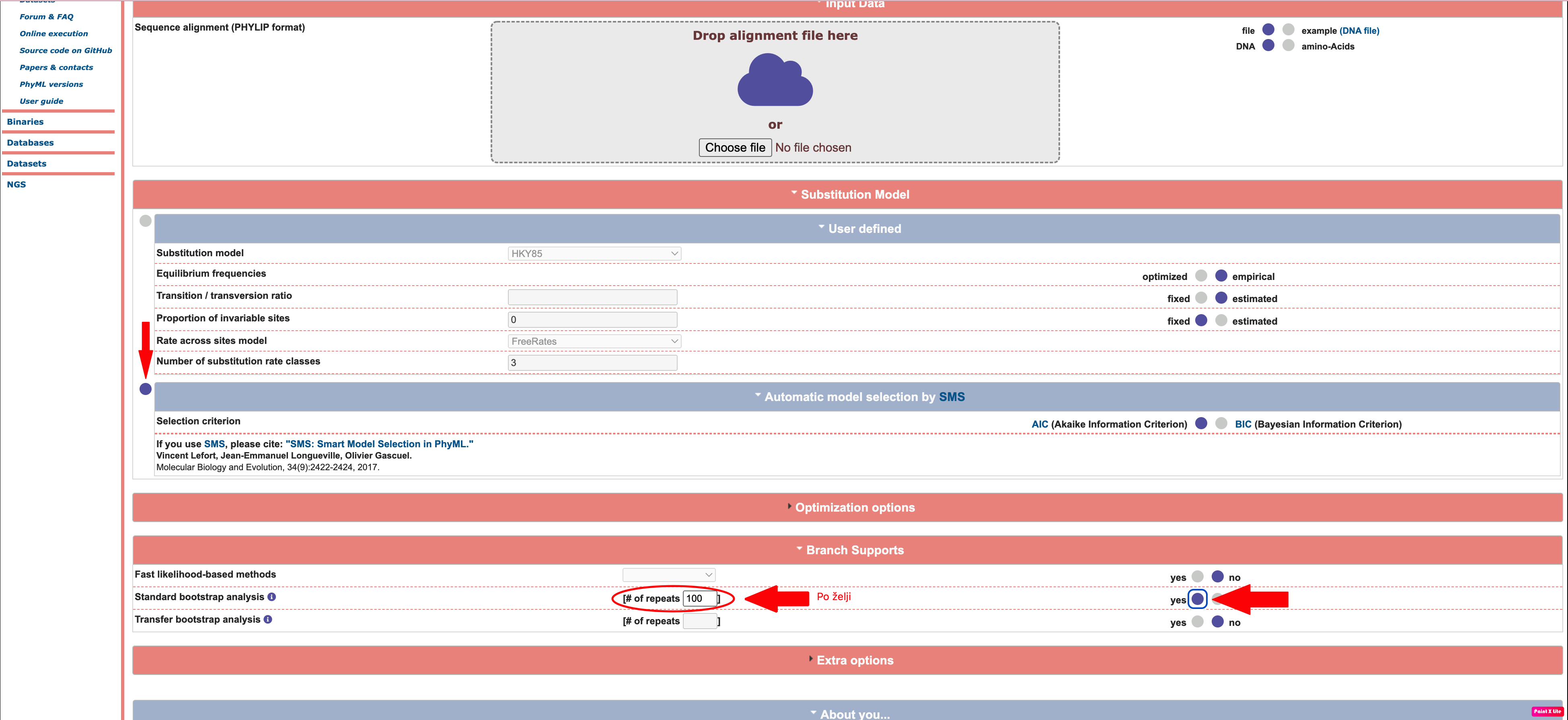
PhyML 3.0 (najnovejša verzija) izvrši nov iskalni algoritem SPR (Subtree-Pruning-Regrafting), ki se zanaša na filter, kateri temelji na varčnosti namesto na razdalji. Ločimo dva algoritma: Multiple_Spr_Cycles in One_Spr_Cycle. Slednji obravnava vsako poddrevo v trenutni filogeniji. Vrednost varčevanja je ocenjena za vsako poddrevo pri vsakem razcepu. Ko dobi vse vrednosti varčevanja, jih razvrsti. Verjetnost za najbolj varčne rešitve so ocenjene s pomočjo uporabe dvostopenjskega pristopa. Na prvi stopnji se oceni verjetnost drevesa po dodatku varčnega SPR koraka brez prilagajanja dolžine vej. Če je dobljena verjetnost večja od do sedaj največje pridobljene verjetnosti, postane novo dobljena verjetnost najboljša. Če dobljena verjetnost ne postane najboljša, pa se ob razcepu tri dolžine vej optimizirajo tako, da povečajo verjetnost. Tukaj je potrebno upoštevati, da so to približne ocene, ki temeljijo na posodobitvi verjetnosti omejenega števila poddreves in ne celotne podatkovne strukture. Ko so vsi SPR koraki ocenjeni, se uporabi največja pridobljena ocenjena verjetnost in celotna podatkovna struktura se popolnoma posodobi. Multiple_Spr_Cycles v prvem koraku obravnava poddrevesa skozi One_Spr_Cycle, nato pa prilagodi parametre nadomestnega modela. Sledi prilagoditev vseh dolžin vej z uporabo metode optimizacije. Zadnji korak je posodobitev celotne podatkovne zbirke. Ti koraki se ponavljajo, dokler se verjetnost ne izboljša.
Vhodni podatki#
Vhodni podatki so aminokislinska oz. nukleotidna zaporedja v formatu Phylip.