TreeFam - podatkovna zbirka dreves živalskih genov#
Avtorja: Špela Sotlar, Jan Trebušak
Datum predstavitve: 2022_05_11
Namen vaje#
Namen vaje je spoznavanje programa TreeFam. To je zbirka genetskih dreves živalskih genomov, uporabljamo pa jo za identifikacijo ortolognih in paralogni genov ter opazovanje poteka evolucije le-teh.
Program#
Program: TreeFam - database of animal gene trees, Release 9
Avtorji programa: Fabian Schreiber, Mateus Patricio, Matthieu Muffato, Miguel Pignatelli, Alex Bateman, vsi delujejo na Sanger Institute (https://www.sanger.ac.uk)
Reference:
Schreiber F, Patricio M, Muffato M, Pignatelli M, Bateman A. TreeFam v9: a new website, more species and orthology-on-the-fly. Nucleic Acids Res. 2014 Jan;42(Database issue):D922-5. doi: 10.1093/nar/gkt1055. Epub 2013 Nov 4. PMID: 24194607; PMCID: PMC3965059
Opis programa#
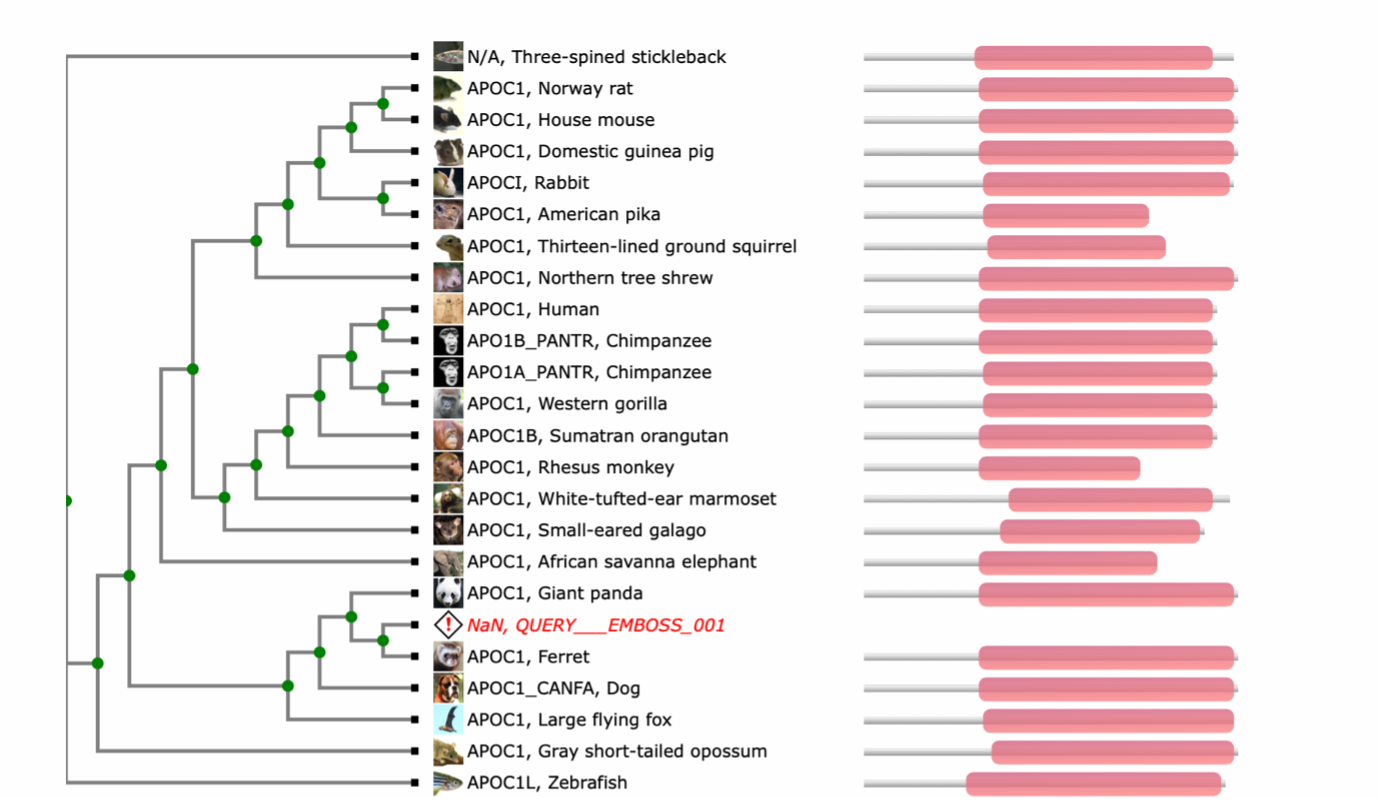
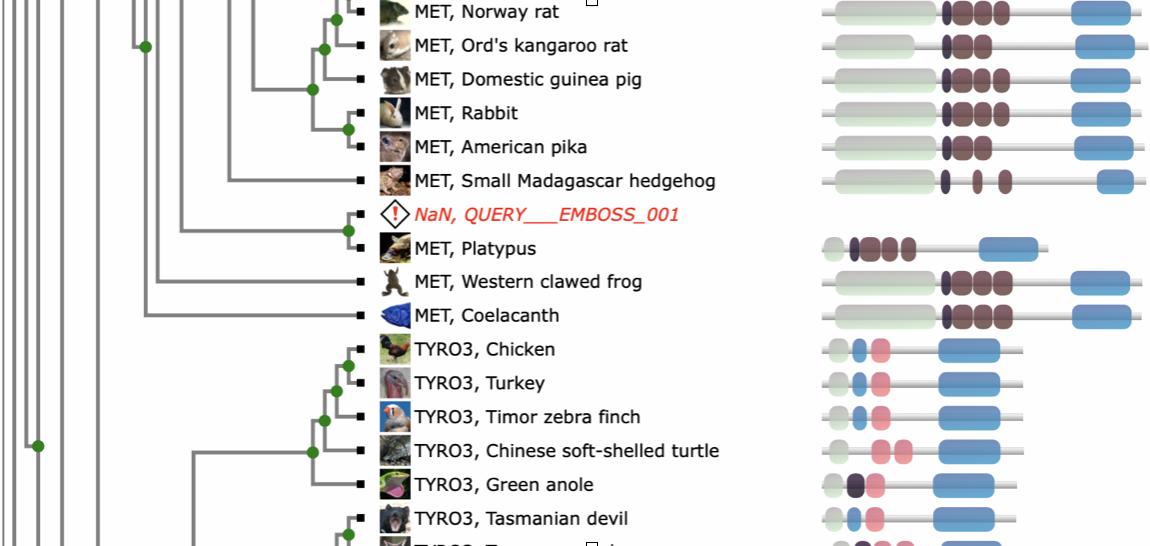
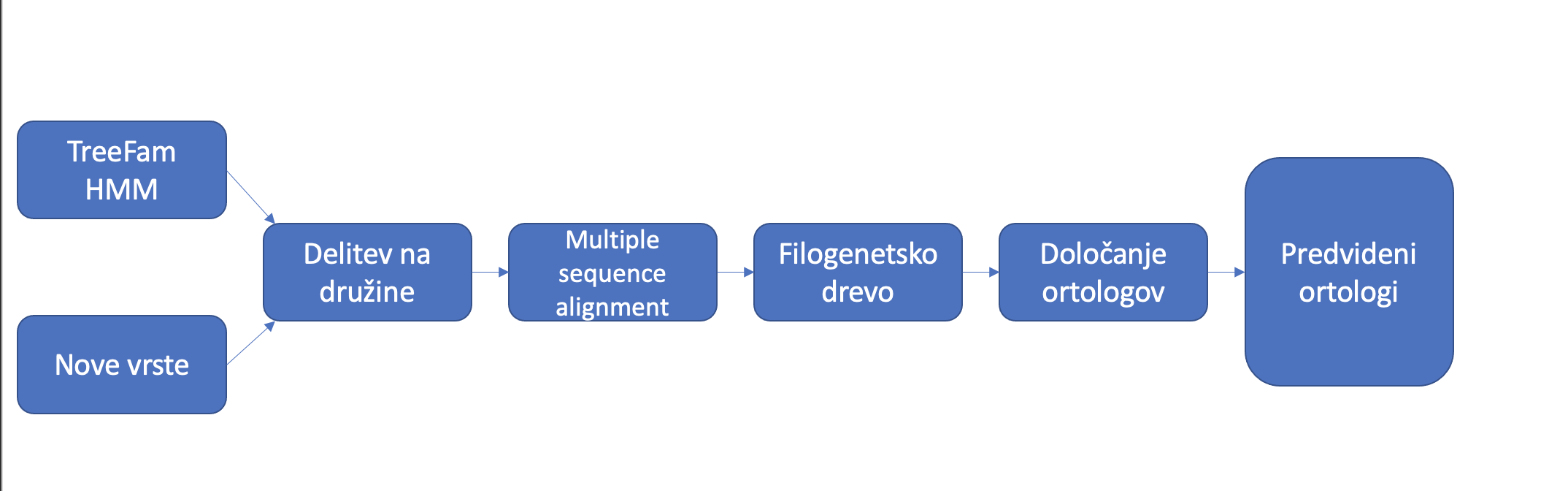
TreeFam je podatkovna zbirka filogenetskih dreves iz živalskih genomov. Zajema 109 vrst in 15736 družin s približno 2,2 milijona zaporedji. Določene baze podatkov napovejo ortologijo/paralogijo glede na filogenetska drevesa, ki so bila skonstruirana z poravnavo homolognih zaporedij – med nje spada tudi TreeFam. Program naredi poravnano zaporedja in ga vmesti v ustrezno družino z uporabo MAFFT (multiple sequence alignment program) nato pa ga doda v ustrezno genetsko drevo z uporabo družine, kot reference za RAxML-EPA algoritem. Prednost filogenetskih dreves pred grafičnimi prikazi je v tem, da si drevesa lažje predstavljamo in so bolj informativna, saj lahko določimo kdaj se je določen gen podvojil/izgubil. TreeFam uporablja Ensembl Compara pipeline, z izjemo definicije genetskih družin. TreeFam uporabi HMM pristop, kar zagotavlja stabilnost družin. Za vsako genetsko družino je poravnava narejena z MCoffee (družine z manj kot 200 člani) oziroma MAFFT. Za vsako družino zgardijo genetsko drevo z uporabo TreeBesta. Ta zgradi pet genetskih dreves glede na aminokislinsko zaporedje. Pet dreves je nato združenih v eno. Prednost uporabe AK zaporedja in kodonskega zaporedja je v tem, da drevesa, ki temeljijo na kodonskem zaporedju razrešijo bližnja sorodstva, glede na AK pa daljna. To skupno drevo je nakoncu združeno s taksonomskim drevesom NCBI z uporabo ‘Duplication/Loss Interference’ algoritma.
Vhodni podatki#
Aminokislinsko zaporedje iskanega proteina v FASTA formatu.