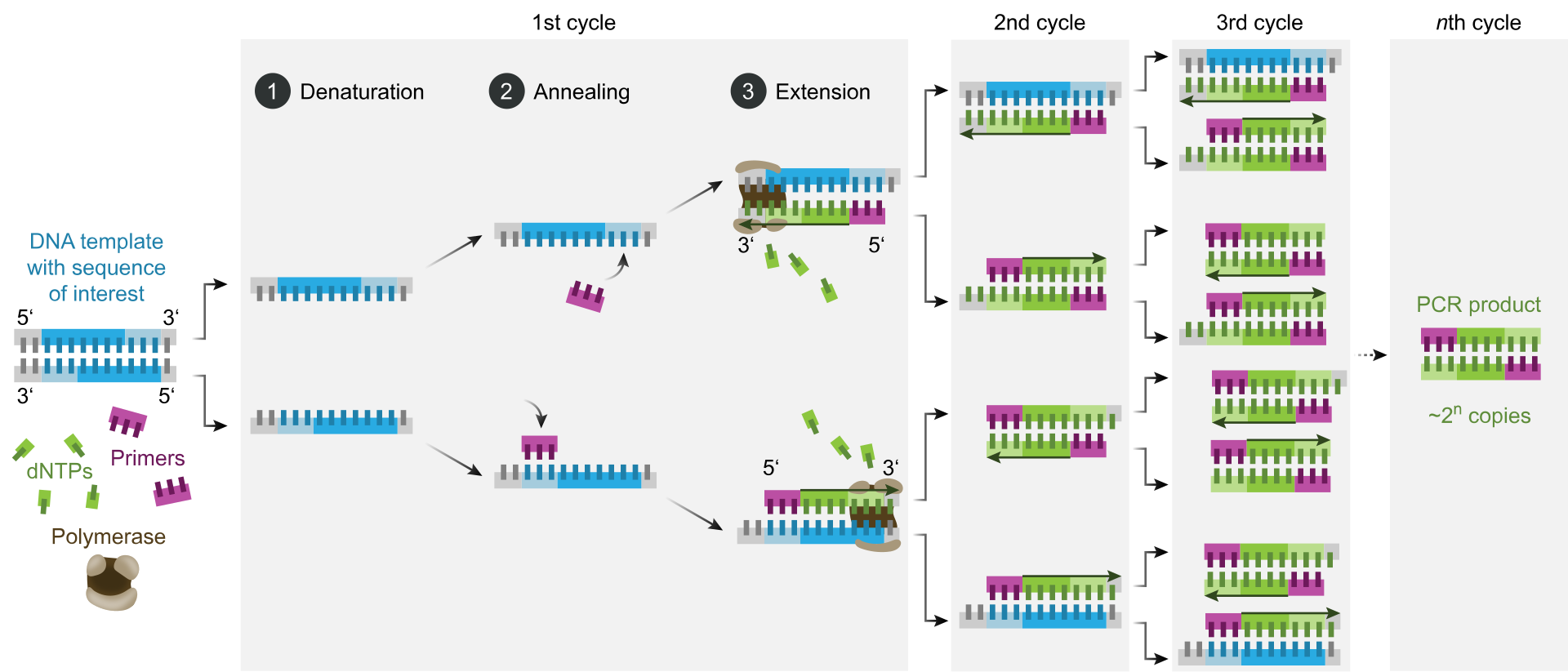
V iskalnik vpišemo NCBI, kot database izberemo Gene in v iskalno vrstico vpišemo Albumin Homo sapiens.
Na desni strani pod zavihkom Related information izberemo RefSeqGene, nato FASTA format in zopet na desni strani Highlight sequence features.
Skrajno levo spodaj lahko izberemo, da nam prikaže eksonske regije celotnega genomskega zaporedja.
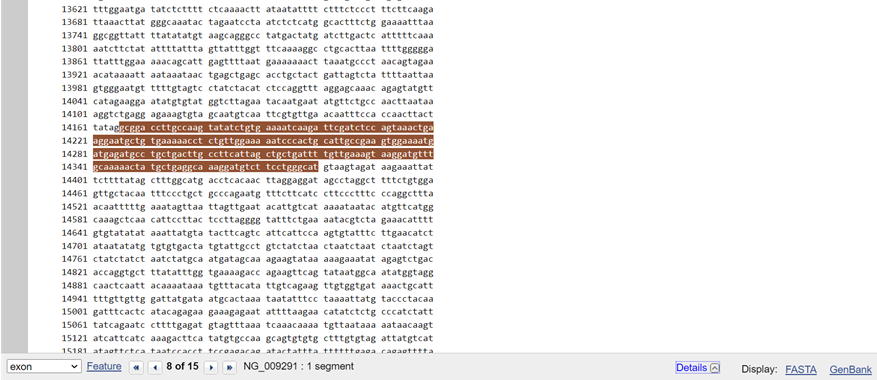
Izberemo si 8. ekson in v desnem spodnjem oknu izberemo FASTA. To je torej zaporedje, ki ga želimo dobiti kot produkt PCR reakcije. Začetni oligonukleotidi (primerji) se morajo vezati med 100. in 50. nukleotidom pred tem zaporedjem. Sam primer je optimalne dolžine med 18 in 24 nukleotidov (če bo prekratek, nam ne bo dal dovolj specifičnih rezultatov, če bo predolg, pa bo faza hibridizacije v PCR reakciji potekala predolgo).
Desno zgoraj pod zavihkom Change region shown povečamo zaporedje pred in po eksonu (8. ekson sega od 14 166. do 14 380. nukleotida).

Pod Selected region povečamo zaporedje tako, da bo segalo od 14 050. do 14 500. nukleotida, nato pa pritisnemo Update view, da nam prikaže novo zaporedje v FASTA formatu.
Na desni strani izberemo Pick primers in preusmeri nas na Primer-BLAST, kjer nam v iskalno okence tudi že kopira pravo NCBI referenčno sekvenco.
Ker želimo, da je med koncem primerja in eksonsko regijo nekaj nukleotidov, na desni strani določimo dolžini primerja - v našem primeru bo začetni (forward primer) segal od 14 050. do 14 120. nukleotida, kar pomeni, da je do začetka eksonske regije približno 50 nukleotidov. Končni (reverse primer) pa naj sega od 14 430. do 14 500. nukleotida.
Malo nižje imamo prikazan zavihek Primer Parameters. Tu prva dva okenca pustimo prazna, izpolnimo ju v primeru, ko imamo že svoje primerje in želimo preveriti le njihovo specifičnost. Pod okencem, imenovanim PCR product size moramo preveriti, če se max usklajuje z našo regijo od začetka forward primerja do konca reverse primerja - v našem primeru regija sega od 14 000. do 14 500. nukleotida, kar pomeni, da lahko max pustimo na 1000.
Melting temperature oz. temperatura tališča (Tm) pove, kdaj bo dsDNA denaturirala do ssDNA. Najbolj optimalne so nastavitve za min. 59 ˚C, opt. 62 ˚C, max. 65 ˚C, max. razlika Tm med obema primerjema pa je 3-5 ˚C.
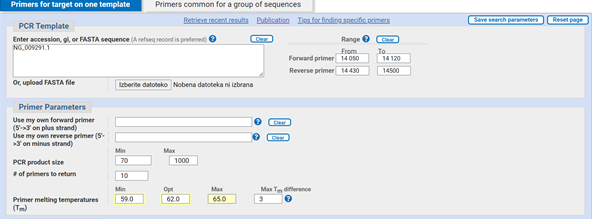
Naslednjega okenca (Exon/intron selection) ne spreminjamo, razen v primeru, da bi v Primer-BLAST kopirali zaporedje z več eksoni. V takem primeru bi morali odkljukati okence, imenovano Intron inclusion, kar bi zagotovilo, da se intron ne bi prepisal. V primeru, da bi se nam intron prepisal, bi dobili podvojeno DNA, česar pa nočemo.
V zadnjem zavihku (Primer Pair Specificity Checking Parameters) lahko vidimo, kateri organizem smo izbrali (9606 označuje Homo sapiens). V tem zavihku ne potrebujemo spreminjati ali izpolnjevati nobenih okenc.
Zdaj smo pregledali vse zavihke in okenca, zato lahko pritisnemo Get primers in počakamo eno do dve minuti.
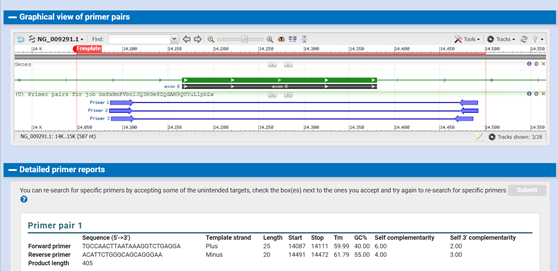
Kot rezultat dobimo seznam ustreznih primerjev, ki jih lahko med seboj primerjamo pod zavihkom Detailed primer reports. Podan je tip primerja, njegova dolžina, njegov začetek in konec, temperatura tališča in delež baznih parov. Ta je najbolj optimalen med 40 % in 60 %, saj zagotavlja dovolj močno vezavo primerja na DNA. Na koncu imamo podano še verjetnost samo-komplementarnosti, pri čemer ne želimo, da se primerja med seboj povežeta ali pa, da se primer veže sam nase. Pogledamo torej, da je self complementarity manj kot 8 in 3’ self complementarity manj kot 5.