HMMER#
Avtorja: Nuša Kos Thaler, Miha Razdevšek
Datum predstavitve: 2022-04-28
Namen vaje#
Namen vaje je s programom HMMER poiskati homologe izbranih proteinov in rezultate primerjati z rezultati pridobljenimi s programom BLAST. Prav tako pa na podlagi primerjav ugotoviti, v katerih primerih je bolje uporabljati HMMER.
Program#
Program: HMMER 3.3.2
Avtorji programa: S.Eddy (HHMI/Harvard Univeristy), R.Finn (EMBL-EMI)
Reference:
Potter, S.C.; Luciani, A.; Park, S.R. Eddy Y.; Lopez, R. and Finn, R.D.;(2018) HMMER web server: 2018 update. Nucleic Acids Research Web Server Issue 46:W200-W204. 10.1093/nar/gky448
Finn, R. D.; Clements, J. and Eddy S. R.; (2011) HMMER web server: interactive sequence similarity searching. Nucleic Acids Research Web Server Issue 39:W29–W37. 10.1093/nar/gkr367
Opis programa#
HMMER je sestavljen iz več programov, ki nam omogočajo poravnavo bioloških zaporedij in iskanje homologov po podatkovnih zbirkah. Uporablja verjetnostni model imenovan profil skritih modelov Markova (Hidden Markov models, HMM). Gre za to, da imamo več možnih stanj sistema (x), ki nam niso poznana (skrita). Poznane so nam pa posledice oz. opažanja, ki so odvisna od skritih stanj. Pomembno je tudi, da je stanje x(n) odvisno samo od stanja x(n-1). Vsi prehodi med stanji in prehodi iz stanj do opažanj imajo neko verjetnost. Algoritem nam na podlagi teh verjetnosti izračuna vsa možna stanja in jih uredi po padajoči verjetnosti. Prednost programa je, da z njim lahko napovemo in poravnamo oddaljene homologe našega proteina.
Program HMMER ima več opcij, ki nam lahko pomagajo pri iskanju homologov. Omočoga tako iskanje homologov posameznih nukleotidnih (nhmmer) in proteinskih (phmmer, jackhmmer) zaporedij z bazami podatkov nukleotidnih in proteinskih zaporedij, kot tudi iskanje homologov pri poravnavah več zaporedij (hmmbuild, hmmalign). V tem seminarju se bova osredotočila na phmmer, kjer proteinsko zaporedje primerjamo z bazo podatkov proteinskih zaporedij.
Podoben program, ki smo ga že spoznali je BLAST. Ta za poravnave uporablja pozicijsko neodvisne substitucijske matrike kot sta BLOSUM in PAM. HMM profil je nadgradnja pozicijsko specifičnih točkovalnih matrik, posebej v smislu pozicijske odvisnosti poravnave vrzeli in insercij. V ozadju programa HMMER se sestavi profil – poravnava večih zaporedij (npr. neke domene proteinske družine). HMMER torej iz našega zaporedja ustvari profil, tako da vrednosti ustrezne matrike (npr. BLOSUM62) spremeni v verjetnosti in doda verjetnost za insercijo ali vrzel. Na podlagi tega nam vrne proteine, ki so našemu zaporedju najbolj podobni.
Vhodni podatki#
Kot vhodne podatke lahko uporabimo:
FASTA format zaporedja proteina
enočrkovno aminokislinsko zaporedje
datoteko, ki vsebuje aminokislinsko zaporedje
kodo za dostop do določenega proteina (accession code)

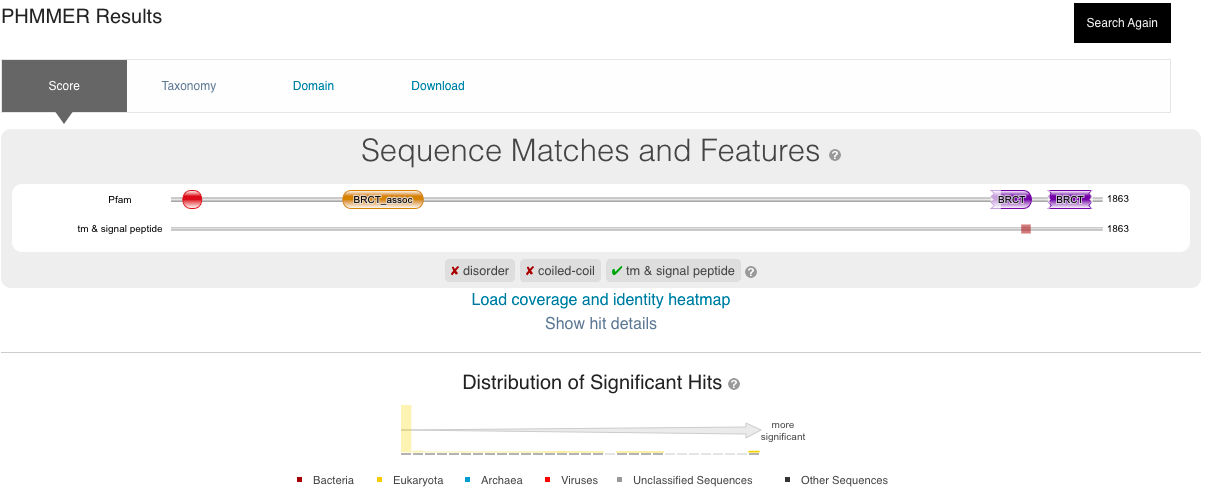
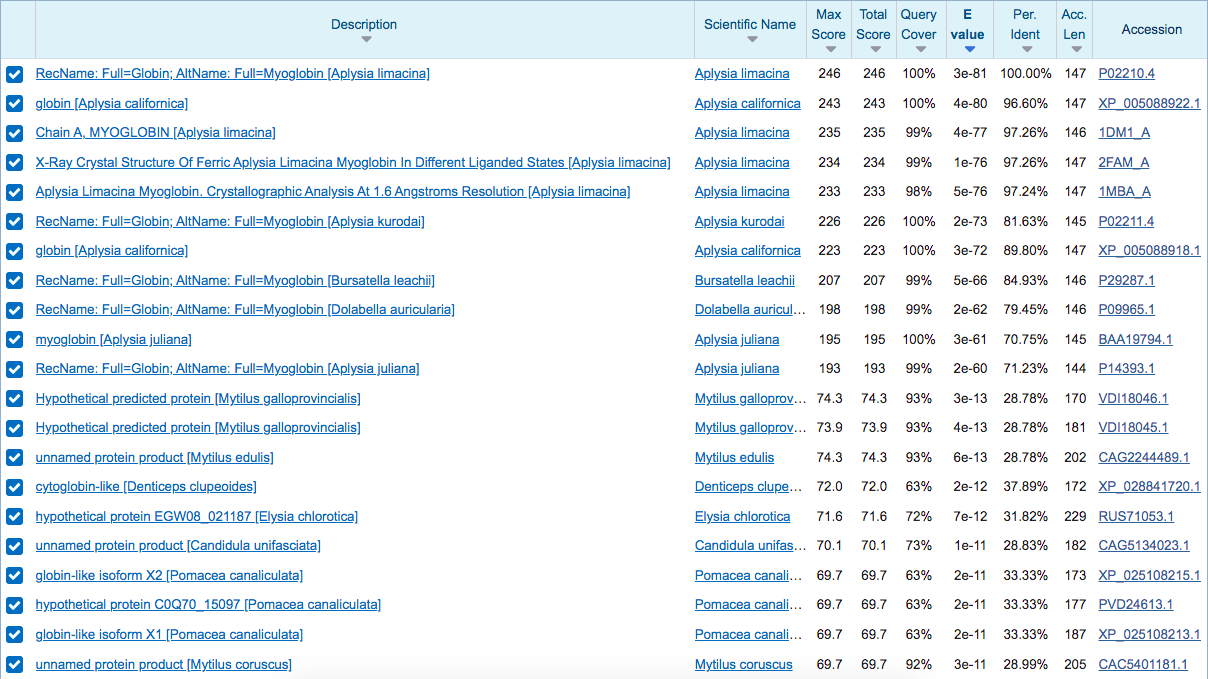
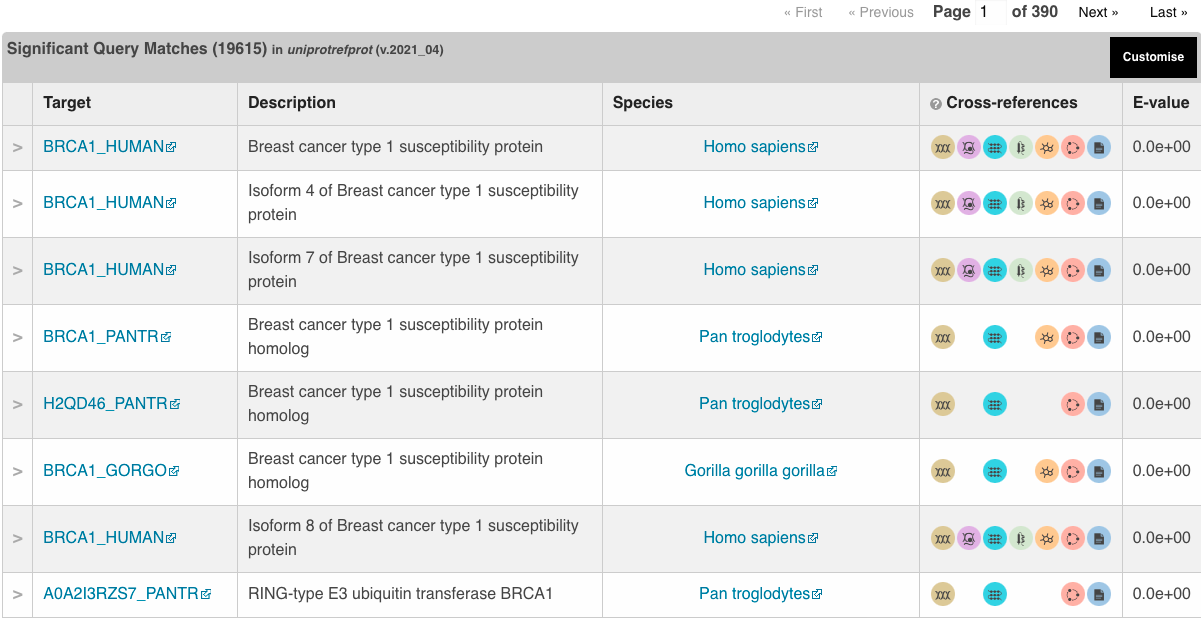 Rezultati programa blastp:
Rezultati programa blastp:
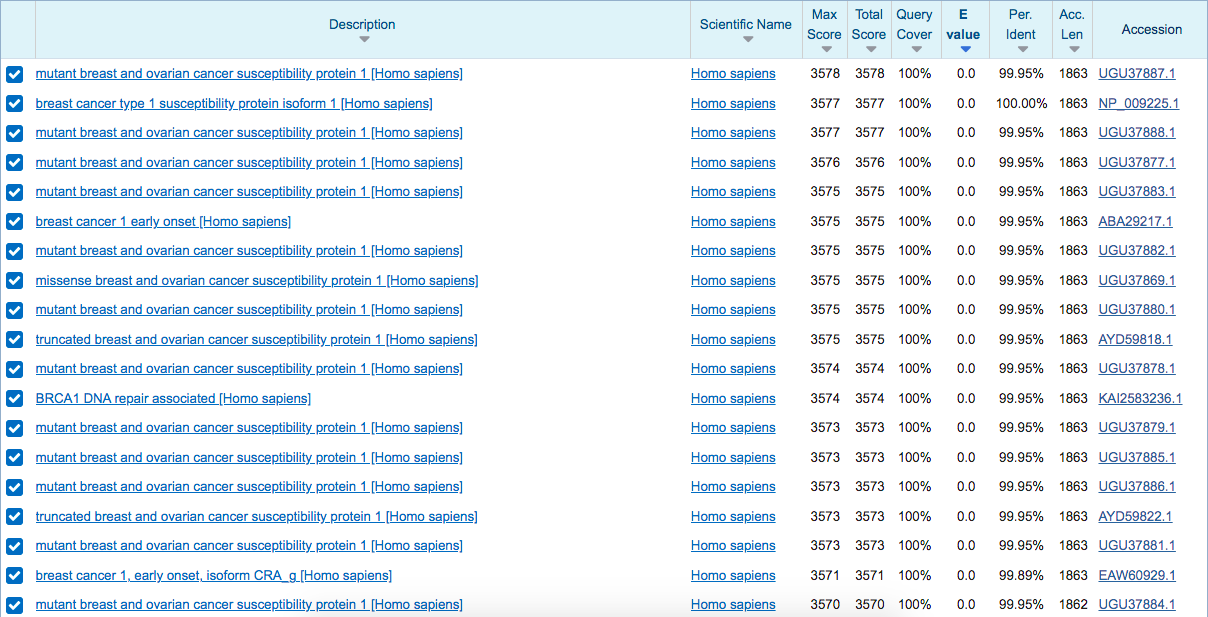
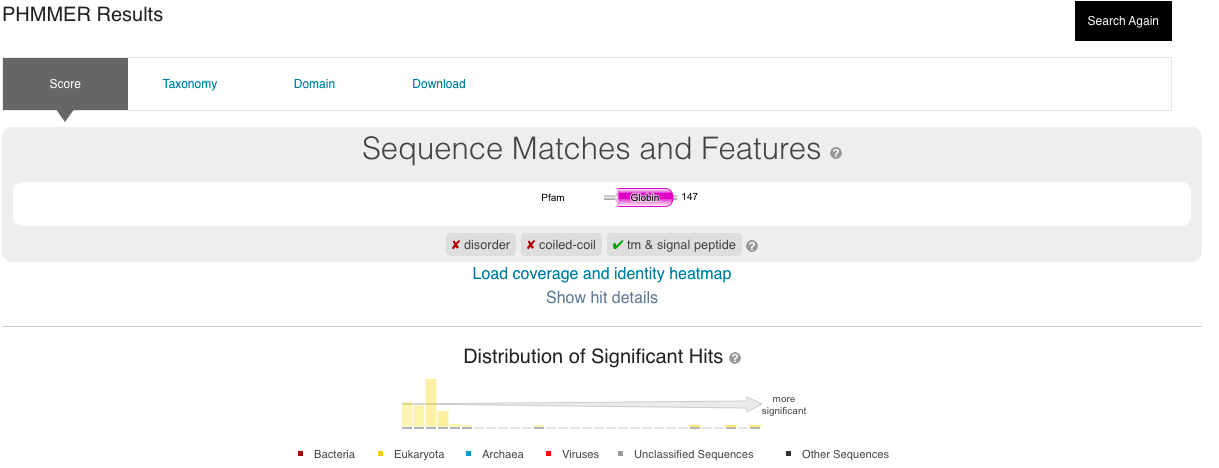
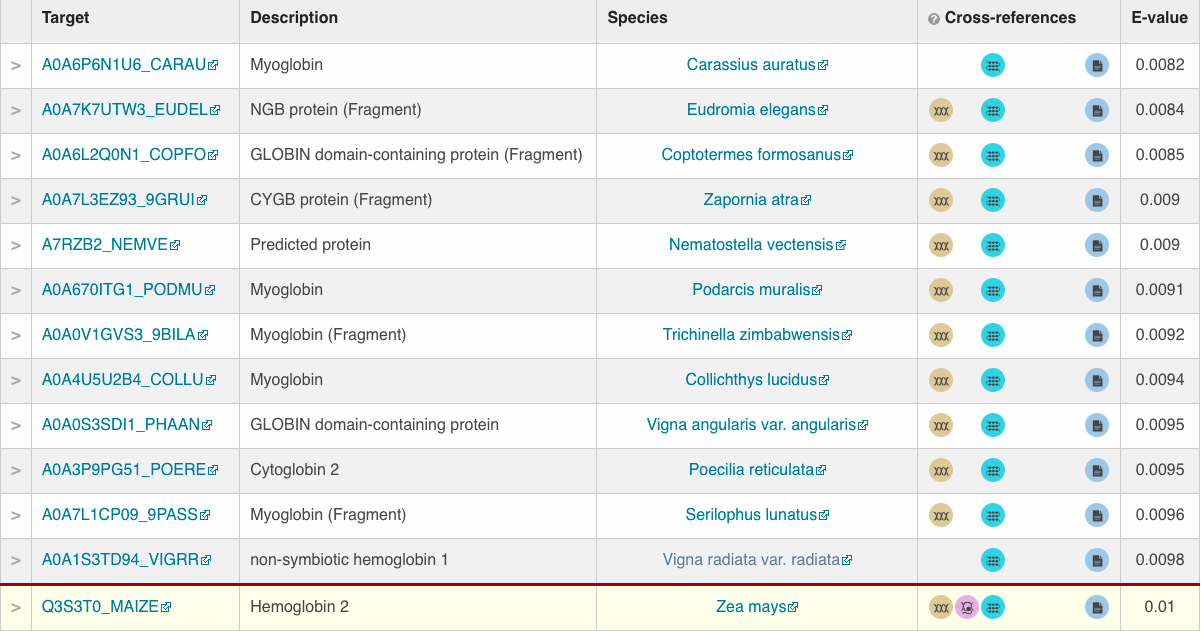 Rezultati programa blastp:
Rezultati programa blastp: