Klasifikacija proteinov z InterPro#
Avtorja: Neža Peternel, Pia Sotlar
Datum predstavitve: 2022-04-20
Namen vaje#
Namen vaje je spoznati osnove uporabe programa InterPro, ki se uporablja za klasifikacijo proteinov v družine in primerjavo različnih lastnosti, kot so domene, iz različnih podatkovnih baz. Pri vaji se bomo naučili interpretirati podatke, ki nam jih program vrne za iskani protein. Seznanili se bomo tudi s funkcijo obratnega iskanja proteinov po domenah in s tem preverili, koliko drugih proteinov vsebuje enake domene kot naš izhodni protein.
Program#
Program: InterPro 88.0
Avtorji programa: Alex Bateman, European Bioinformatics Institute, Cambridge, UK
Reference:
Blum, M.; Chang, H.; Chuguransky, S.; Grego, T.; Kandasaamy, S.; Mitchell, A.; Nuka, G.; Paysan-Lafosse, T.; Qureshi, M.; Raj, S.; Richardson, L.; Salazar, G.A.; Williams, L.; Bork, P.; Bridge, A.; Gough, J.; Haft, D.H.; Letunic, I.; Marchler-Bauer, A.; Mi, H.; Natale, D.A.; Necci, M.; Orengo, C.A.; Pandurangan, A.P.; Rivoire, C.; Sigrist, C.J.A.; Sillitoe, I.; Thanki, N.; Thomas, P.D.; Tosatto, S.C.E.; Wu, C.H.; Bateman, A. and Finn, R.D. (2020) The InterPro protein families and domains database: 20 years on. Nucleic Acids Research. 10.1093/nar/gkaa977
Opis programa#
InterPro je sekundarna podatkovna zbirka, ki zbira in obdeluje podatke iz trinajstih različnih podatkovnih baz (npr. CATH, PANTHER, PFAM). Ker pa večina od teh baz črpa podatke direktno iz primarnih podatkovnih baz (npr. UniProt), lahko rečemo, da je InterPro tudi terciarna podatkovna zbirka. Na podlagi podatkov iz prej omenjenih podatkovnih baz InterPro analizira proteinska zaporedja. Ker vsaka se vsaka podatkovna baza osredotoča na določeno klasifikacijo, InterPro te podatke zbere in posamezne proteine razporeja v družine ter napoveduje domene in različna pomembna mesta v samem proteinu. Proces je prikazan na spodnji shemi:
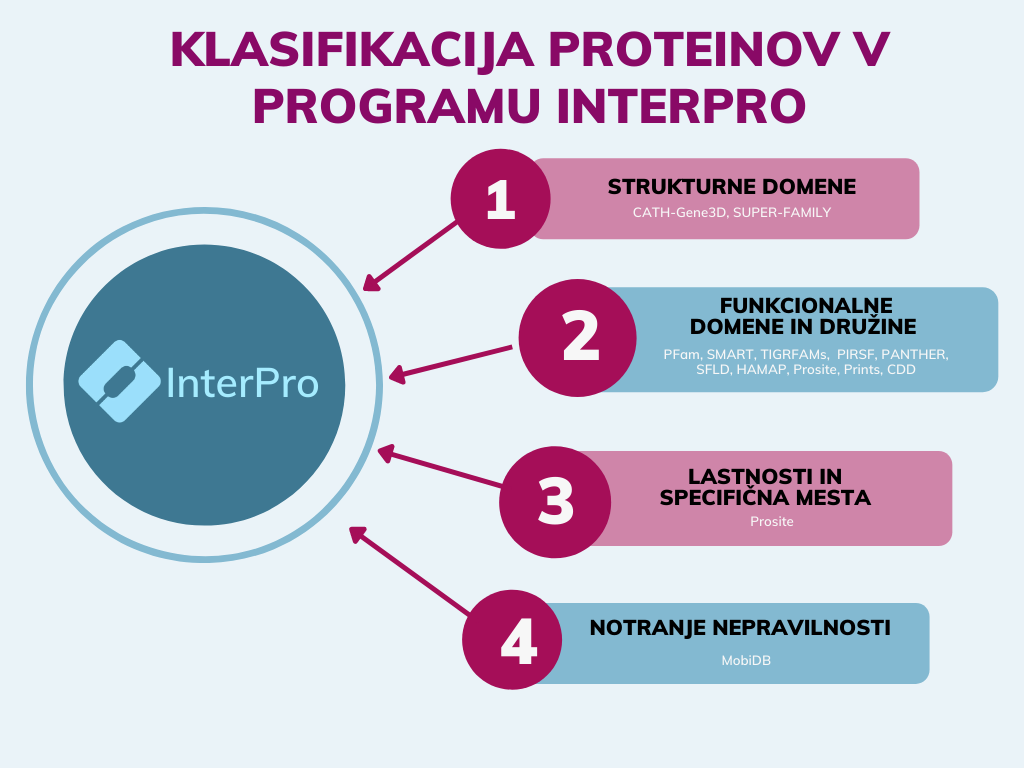
Rezultate prikaže v shematski obliki, kjer so združeni podatki iz različnih dostopnih podatkovnih baz, kar omogoča neposredno primerjavo klasifikacije iskanega proteina. Med rezultati najdemo družine, domene, homologne superdružine, ponavljajoče regije, aktivna in vezavna mesta. Posebna prednost programa so tudi t.i. gensko ontološki termini, ki ponujajo hiter vpogled v samo funkcijo proteina, sodelovanje pri bioloških procesih in morebitno nahajanje v različnih celičnih organelih. Primer izpisa rezultatov je prikazan spodaj:
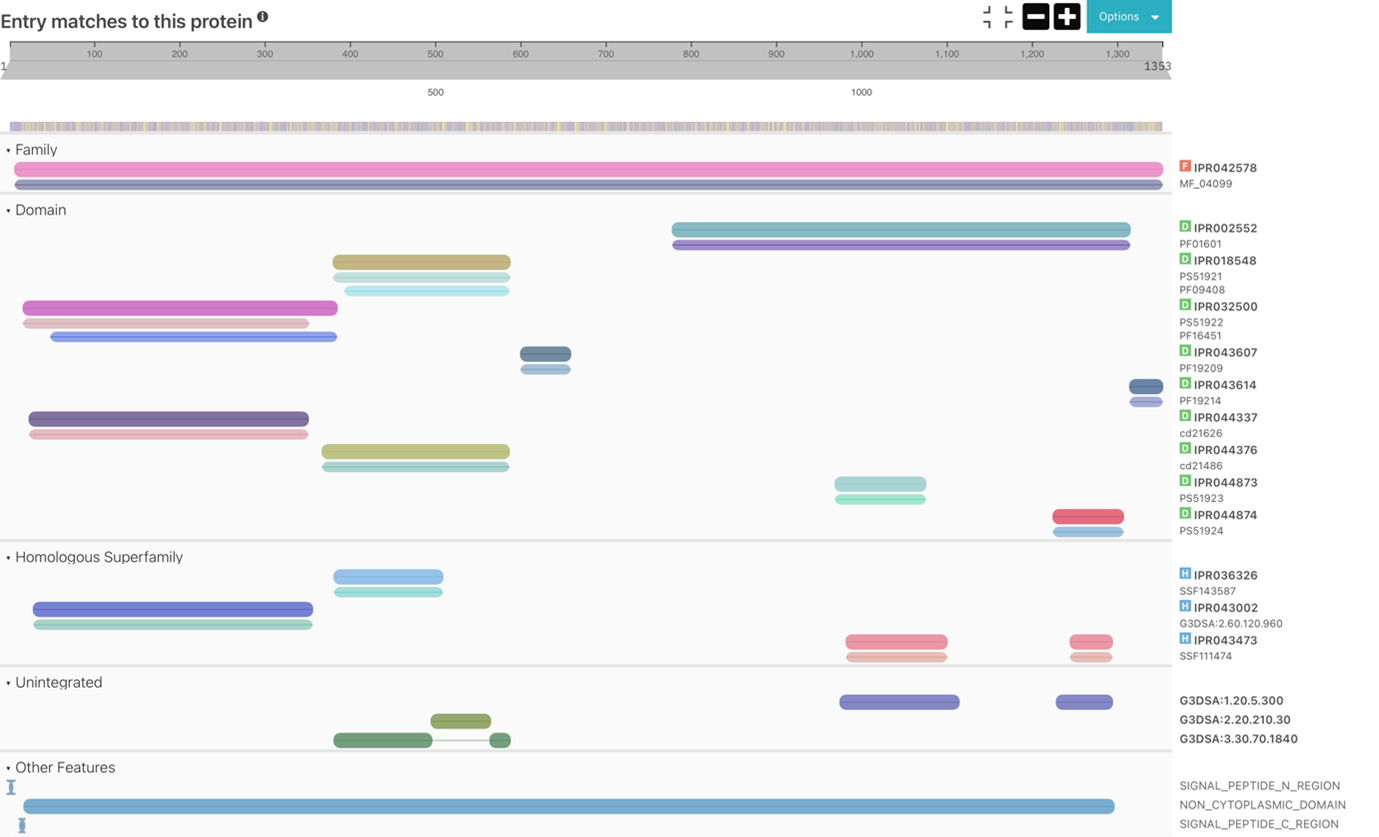

Pri nekaterih proteinih program ponuja tudi iskanje podobnih proteinov in s pomočjo programa AlphaFold izriše predvideno zvitje proteina.
Program je primeren bolj ali manj za vsa aminokislinska zaporedja različnih organizmov, saj deluje na podlagi široke palete podatkovnih baz, ki vsebujejo podatke o najrazličnejših lastnostih proteinov.
Vhodni podatki#
Iskanje lahko poteka na več različnih načinov:
z navedbo aminokislinskega zaporedja v FASTA formatu,
preko kode za dostop določenega proteina,
preko imena družine, domene, ali super družine,
preko ključnih besed ali GO terminov,
preko nahajanja domen.
