NetSolP - topnost proteinov
Avtorja: Pia Trošt, Tina Zajec
Datum predstavitve: 2022-04-20
Namen vaje
Izražanje proteinov v sistemu E. coli za potrebe biotehnologije, farmacevtske industrije in zadnje čase vse bolj tudi medicine, je zaradi enostavnosti, cenovne ugodnosti in predvsem hitrosti postalo precej priljubljeno. Pogosto pa napačno zvitje in tvorba inkluzijskih telesc, onemogočajo in močno zmanjšujejo izkoristek takšne proizvodnje. V namen povečanja topnosti proteinskih produktov je poleg prilagajanja pogojev eksperimenta precej priročna tudi manipulacija aminokislinskega zaporedja. Pri načrtovanju in optimizaciji le-te, oziroma predvidevanju topnosti proteina na podlagi aminokislinskega zaporedja si lahko pomagamo z obravnavanim programom.
Program
Program: NetSolP - 1.0
Avtorji programa: Vineet Thumuluri, Hannah-Marie Martiny (Research Group for Genomic Epidemiology, National Food Institute, Technical
University of Denmark), Jose J. Almagro Armenteros (Faculty of Health and Medical
Sciences, University of Copenhagen), Jesper Salomon (Novozymes A/S), Henrik Nielsen (Department of Health Technology, Technical University of Denmark), Alexander R. Johansen (Department of Computer Science, Stanford University)
Reference:
Thumuluri, V.; Martiny, H.M.; Armenteros, J.J.A; Salomon, J.; Nielsen, H.; Rosenberg Johansen, A. (2021) NetSolP: predicting protein solubility in Escherichia coli using language models. Bioinformatics 38, 941–946. 10.1093/bioinformatics/btab801
Opis programa
Program NetSolP-1.0 s kompleksnim modelom procesiranja naravnega jezika - seveda tukaj prilagojenega na jezik proteinov - iz aminokislinskega zaporedja predvidi topnost in uporabnost proteinov v ekspresijskem sistemu E.coli. Globoko učenje na modelu transformerjev omogoča, da posamezne aminokisline postavi v kontekst celotnega proteina in se tako na tej višji ravni osredotoča na tiste, ki so pomembnejše za določanje topnosti in eksperimentalne uporabnosti našega iskalnega niza. Profil iskalnega niza vzpostavi s primerjavo s podatki iz podatkovnih zbirk na katerih je program “naučen” (podatkovna zbirka PSI: Biology dataset, podatkovna zbirka Price inštituta North East Structural Consortium, zbirka Camsol z eksperimentalnimi podatki o 19 proteinih s 56 mutacijskimi variantami).
Zaradi velike količine podatkov, ki jih program pri svoji izvedbi uporabi in omejitve velikosti transformerskega modela, program naenkrat obravnava zgoj 1022 aminokislin, posledično je njegova napoved boljša in seveda tudi hitrejša za krajše iskalne nize.
Vhodni podatki
Vhodni podatki so eno ali več aminokislinskih zaporedij v FASTA formatu.
Navodila
Vhodni podatki
Vhodni podatki za 1.primer:
aminokislinsko zaporedje človeškega hemoglobina (RCSB PDB ID 1A00)
aminokislinsko zaporedje mišjega globina Peromyscus maniculatus (RCSB PDB ID 4H2L)
aminokislnsko zaporedje hemoglobina Mammuthus primigenius (RCSB PDB ID 3VRG)
aminokislinsko zaporedje cianoglobina cianobakterije Synechocystis sp. (RCSB PBD ID 1S69)
aminokislinsko zaporedje konjskega hemoglobina verige alfa (UniProt ID P01958) in beta (UniProt ID P02062)
urejena datoteka z zaporedji
Vhodni podatki za 2. primer:
aminokislinsko zaporedje človeškega EpCAM (UniProt ID P16422)
aminokislinsko zaporedje človeškega albumina (UniProt ID P02768)
aminokislinsko zaporedje človeške gama podenote receptorja za interlevkin 2 (IL2RG) (UniProt ID P31785)
aminokislinsko zaporedje človeškega aktina (UniProt ID P60709)
aminokislinsko zaporedje človeškega z G-proteinom sklopljenega receptorja 3 (GPR3) (UniProt ID P46089)
Postopek dela
naloga:
V iskalno okence programa NetSolP vstavite aminokislinska zaporedja homologov hemoglobina sesalcev in cianoglobina predstavnika cianobakterij, spodaj pod okncem označite kot kaže slika : 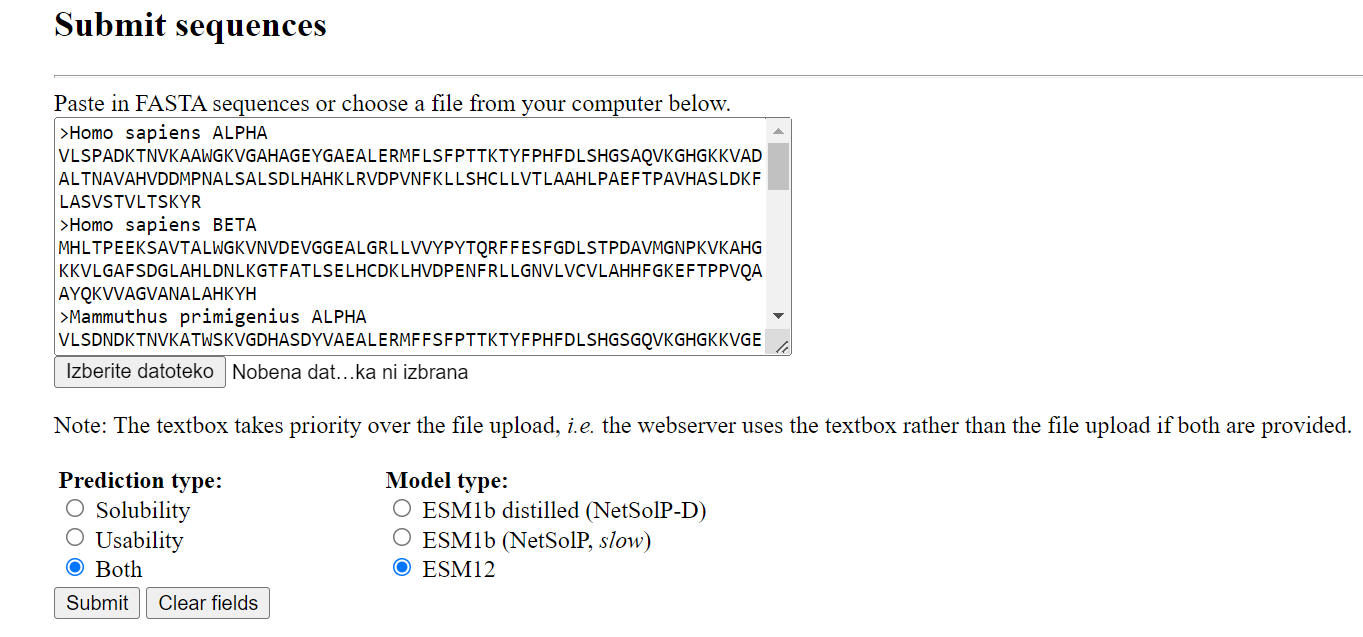 Po podatkih v članku naj bi bila nastavitev ESM12 primernejša za primerjavo podobnih zaporedij.
Po podatkih v članku naj bi bila nastavitev ESM12 primernejša za primerjavo podobnih zaporedij.
Kaj nam dobljeni rezultati povedo o topnosti in uporabnosti zgoraj omenjenih proteinov v ekspresijskem sistemu E.coli? Ali bi bilo izražanje konjskega hemoglobina v E.coli smiselno?
naloga:
V iskalno okence programa NetSolP vstavite aminokislinska zaporedja za 2. primer, spodaj pod okncem označite kot kaže slika: 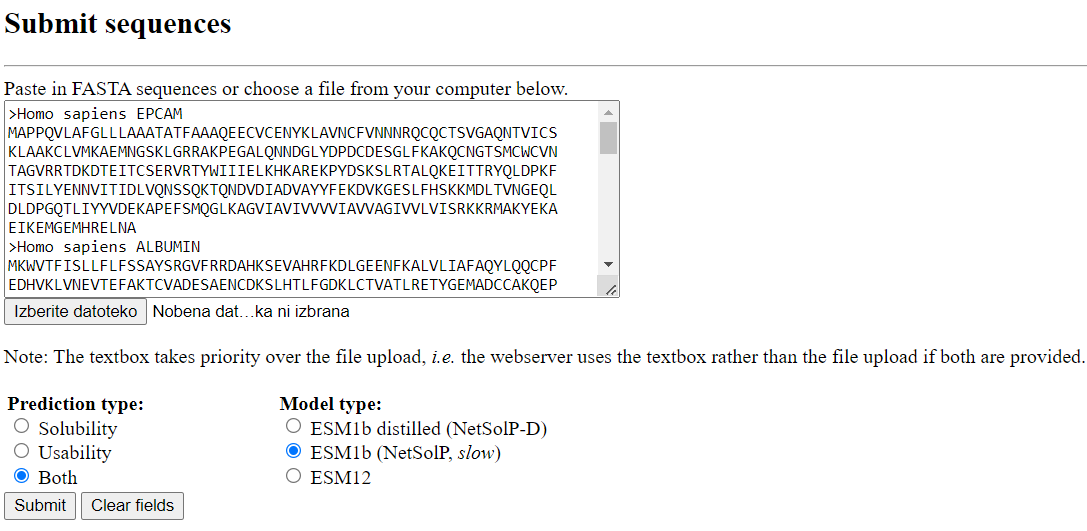
Pričakovani rezultati in razlaga
Po vnosu iskalnega niza 1. naloge bi nam program moral izpisati nekaj podobnega kot prikazuje spodnja slika:
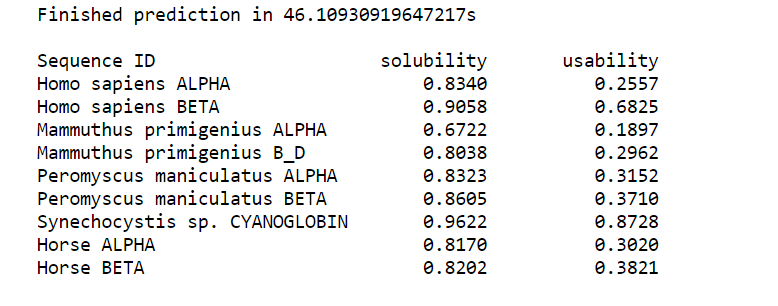
Že po kodi zapisa za prve štiri primere proteinov lahko ugotovimo, da smo si datoteke FASTA sposodili iz zbirke RCSB PDB, ki vsebuje eksperimentalno razrešene strukture proteinov. Če bi se še nekoliko potopili v pripise, bi ugotovili, da je za vse štiri proteine ekspresijski organizem E.coli. Pričakovano je napovedana topnost za te štiri proteine (oz. njihove verige) visoka. Za razliko od človeškega, mamutovega, mišjega in cianobakterijskega homologa pa zbirka PDB ne vsebuje strukture konjskega hemoglobina. Glede na siceršnjo evolucijsko sorodnost (med sesalci seveda) in po ocenitvi rezultata NetSolP lahko predvidimo, da bi bilo njegovo izražanje ravno tako možno z ekspresijo v E.coli.
Vprašanje v razmislek:
Zakaj je vrednost uporabnosti pri cianoglobinu tako visoka?
Po vnosu iskalnega niza 2. naloge bi nam program moral izpisati nekaj podobnega kot prikazuje spodnja slika: 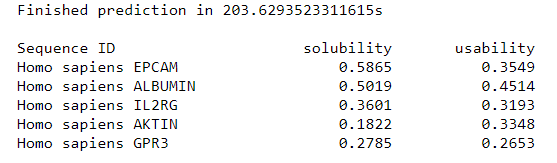
Za razliko od proteinov v 1. nalogi dobimo tukaj precej manjše topnosti in uporabnosti. Problem je lahko nezmožnost E. coli izvajanja posttranslacijskih modifikacij, kar se kaže na primeru EpCAM, albumina in gama podenote receptorja za interlevkin 2. Ti namreč vsebujejo disulfidne vezi in mesta glikozilacije. K netopnosti pa prispeva tudi hidrofobnost proteinov, kar se kaže na primeru gama podenote receptorja za interlevkin 2 in z G-proteinom sklopljenega receptorja 3. Oba sta namreč vsidrana v celično membrano. Aktin pa je značilen le za evkariontske celice in je zato njegovo izražanje v E. coli toliko slabše.

 Po podatkih v članku naj bi bila nastavitev ESM12 primernejša za primerjavo podobnih zaporedij.
Po podatkih v članku naj bi bila nastavitev ESM12 primernejša za primerjavo podobnih zaporedij.

